-
框架核酸是核酸分子通过自组装形成的一维到三维的框架结构, 不仅能精准定位功能基元, 还可实现在纳米甚至原子级尺度上进行力学、光学和电学等物理性质, 以及单分子水平化学与生化反应的精准调控. 利用框架核酸对物质进行原子级的人工自组装, 可实现基本构筑单元的精准物理排布与功能化集成, 进而实现器件制造, 有望推动从原子到宏观的精确功能化的制备. 本文围绕框架核酸和原子制造两大前沿的交叉领域, 阐述框架核酸在原子级精准构筑方面的可行性和优势, 首先介绍了具有原子级精准性的框架核酸的构建, 以及利用框架核酸进行功能化组装的一般策略, 然后着重探讨框架核酸在器件构筑方面的研究进展, 最后就面向原子制造的未来发展方向进行了展望.In recent years, the technology of traditional integrated circuit fabrication is facing a huge challenge. As the top-down lithography gradually approaches to its size limit, the development of atomic-scale precise fabrication for functional devices has already become a major scientific issue at present and might become a breakthrough in the development of information technology in the future. With the reference of the bottom-up self-assembly, which is the basic principle of constructing various advanced structures in living systems, the integrated assembly of atoms can be gradually constructed through a series of operations such as capturing, positioning, and moving atoms. The advent of framework nucleic acids (FNAs) happens to provide a new platform for manipulating single atom or integrating multiple atoms. As is well known, the nucleic acids are not only the carriers of genetic information, but also biological building blocks for constructing novel microscopic and macroscopic materials. The FNAs represent a new type of framework with special properties and features, constructed by nucleic acids’ bottom-up self-assembly. With the improvement of chemical synthesis and modification method of nucleic acids, various molecules and materials, such as fluorophores, nanoparticles, proteins, and lipids, can be spatially organized on FNAs with atomic precision, and these functionalized FNAs have been widely explored in the fields of biosensing, biocomputing, nano-imaging, information storage, nanodevices, etc. Based on the features of precise addressability, superior programmability and tailorable functionality, FNAs can be used for implementing the artificial self-assembly of objects with atomic precision to realize the precise arrangement in spatial and functional integration of basic assembly units, and even prompt the development of device fabrication from atomic scale to macroscopic scale. This review focuses on the intersection of FNAs and atomic fabrication, giving a systematically description of the feasibility and advantages of precisely atomic fabrication with FNAs from three aspects. First, the DNA/RNA nanoarchitectures from static state to dynamic state and general strategies for programmable functionalization of FNAs are briefly introduced. Then the applications of FNAs in device fabrication are highlighted, including single molecule reactors, single molecule sensors, nanodevices for cargo loading and transporting, nanophotonics, nanoelectronics and information processing devices. Finally, an outlook of the future development of atomic fabrication with FNAs is given as well.
-
Keywords:
- framework nucleic acids /
- atomic fabrications /
- self-assembly /
- functional devices
[1] Chang C Z, Zhang J, Feng X, et al. 2013 Science 340 167
 Google Scholar
Google Scholar
[2] Yu R, Zhang W, Zhang H J, Zhang S C, Dai X, Fang Z 2010 Science 329 61
 Google Scholar
Google Scholar
[3] Li Y 2018 Phys. Rev. A 98 012336
 Google Scholar
Google Scholar
[4] Fei H, Dong J, Arellano-Jiménez M J, Ye G, Dong Kim N, Samuel E L G, Peng Z, Zhu Z, Qin F, Bao J, Yacaman M J, Ajayan P M, Chen D, Tour J M 2015 Nat. Commun. 6 8668
 Google Scholar
Google Scholar
[5] Qiao B, Wang A, Yang X, Allard L F, Jiang Z, Cui Y, Liu J, Li J, Zhang T 2011 Nat. Chem. 3 634
 Google Scholar
Google Scholar
[6] Eigler D M, Schweizer E K 1990 Nature 344 524
 Google Scholar
Google Scholar
[7] Crommie M F, Lutz C P, Eigler D M 1993 Science 262 218
 Google Scholar
Google Scholar
[8] Hänsel W, Hommelhoff P, Hänsch T W, Reichel J 2001 Nature 413 498
 Google Scholar
Google Scholar
[9] Feynman R P 1960 Eng. Sci. 23 22
[10] Suess B, Weigand J E 2008 RNA Biol. 5 24
 Google Scholar
Google Scholar
[11] Maurel M C, Leclerc F, Hervé G 2020 Chem. Rev. 120 4898
 Google Scholar
Google Scholar
[12] Chen L L, Yang L 2015 RNA Biol. 12 381
 Google Scholar
Google Scholar
[13] Parkinson G N, Lee M P H, Neidle S 2002 Nature 417 876
 Google Scholar
Google Scholar
[14] Day H A, Pavlou P, Waller Z A E 2014 Bioorg. Med. Chem. 22 4407
 Google Scholar
Google Scholar
[15] Jones M R, Seeman N C, Mirkin C A 2015 Science 347 1260901
 Google Scholar
Google Scholar
[16] 樊春海, 刘冬生 2011 DNA纳米技术: 分子传感、计算与机器 (北京: 科学出版社) 第19页
Fan C, Liu D 2011 DNA Nanotechnology: Moelcular Sensoring, Computation and Machines. (Beijing: Science Press) p19 (in Chinese)
[17] Winfree E, Liu F, Wenzler L A, Seeman N C 1998 Nature 394 539
 Google Scholar
Google Scholar
[18] Yan H, Park S H, Finkelstein G, Reif J H, LaBean T H 2003 Science 301 1882
 Google Scholar
Google Scholar
[19] Lin C, Liu Y, Rinker S, Yan H 2006 ChemPhysChem 7 1641
 Google Scholar
Google Scholar
[20] Goodman R P, Schaap I A T, Tardin C F, Erben C M, Berry R M, Schmidt C F, Turberfield A J 2005 Science 310 1661
 Google Scholar
Google Scholar
[21] He Y, Ye T, Su M, Zhang C, Ribbe A E, Jiang W, Mao C 2008 Nature 452 198
 Google Scholar
Google Scholar
[22] Rothemund P W K 2006 Nature 440 297
 Google Scholar
Google Scholar
[23] Ge Z, Gu H, Li Q, Fan C 2018 J. Am. Chem. Soc. 140 17808
 Google Scholar
Google Scholar
[24] Wang F, Zhang X, Liu X, Fan C, Li Q 2019 Small 15 1900013
 Google Scholar
Google Scholar
[25] Song X, Reif J 2019 ACS Nano 13 6256
 Google Scholar
Google Scholar
[26] Yang F, Li Q, Wang L, Zhang G J, Fan C 2018 ACS Sens. 3 903
 Google Scholar
Google Scholar
[27] Hu Q, Li H, Wang L, Gu H, Fan C 2019 Chem. Rev. 119 6459
[28] Seeman N C 1982 J. Theor. Biol. 99 237
 Google Scholar
Google Scholar
[29] Kuzuya A, Wang R, Sha R, Seeman N C 2007 Nano Lett. 7 1757
 Google Scholar
Google Scholar
[30] Ding B, Sha R, Seeman N C 2004 J. Am. Chem. Soc. 126 10230
 Google Scholar
Google Scholar
[31] He Y, Chen Y, Liu H, Ribbe A E, Mao C 2005 J. Am. Chem. Soc. 127 12202
 Google Scholar
Google Scholar
[32] Chen J, Seeman N C 1991 Nature 350 631
 Google Scholar
Google Scholar
[33] Zheng J, Birktoft J J, Chen Y, Wang T, Sha R, Constantinou P E, Ginell S L, Mao C, Seeman N C 2009 Nature 461 74
 Google Scholar
Google Scholar
[34] Guo P 2010 Nat. Nanotechnol. 5 833
 Google Scholar
Google Scholar
[35] Afonin K A, Bindewald E, Yaghoubian A J, Voss N, Jacovetty E, Shapiro B A, Jaeger L 2010 Nat. Nanotechnol. 5 676
 Google Scholar
Google Scholar
[36] Severcan I, Geary C, Chworos A, Voss N, Jacovetty E, Jaeger L 2010 Nat. Chem. 2 772
 Google Scholar
Google Scholar
[37] 钱璐璐, 汪颖, 张钊, 赵健, 潘敦, 张益, 刘强, 樊春海, 胡钧, 贺林 2006 科学通报 51 2860
 Google Scholar
Google Scholar
Qian L, Wang Y, Zhang Z, Zhao J, Pan D, Zhang Y, Liu Q, Fan C, Hu J, He L 2006 Chin. Sci. Bull. 51 2860
 Google Scholar
Google Scholar
[38] Andersen E S, Dong M, Nielsen M M, Jahn K, Subramani R, Mamdouh W, Golas M M, Sander B, Stark H, Oliveira C L P, Pedersen J S, Birkedal V, Besenbacher F, Gothelf K V, Kjems J 2009 Nature 459 73
 Google Scholar
Google Scholar
[39] Douglas S M, Dietz H, Liedl T, Högberg B, Graf F, Shih W M 2009 Nature 459 414
 Google Scholar
Google Scholar
[40] Han D, Pal S, Yang Y, Jiang S, Nangreave J, Liu Y, Yan H 2013 Science 339 1412
 Google Scholar
Google Scholar
[41] Benson E, Mohammed A, Gardell J, Masich S, Czeizler E, Orponen P, Högberg B 2015 Nature 523 441
 Google Scholar
Google Scholar
[42] Zhang F, Jiang S, Wu S, Li Y, Mao C, Liu Y, Yan H 2015 Nat. Nanotechnol. 10 779
 Google Scholar
Google Scholar
[43] Han D, Pal S, Nangreave J, Deng Z, Liu Y, Yan H 2011 Science 332 342
 Google Scholar
Google Scholar
[44] Williams S, Lund K, Lin C, Wonka P, Lindsay S, Yan H 2009 LNCS 5347 90
[45] Douglas S M, Marblestone A H, Teerapittayanon S, Vazquez A, Church G M, Shih W M 2009 Nucl. Acids Res. 37 5001
 Google Scholar
Google Scholar
[46] Veneziano R, Ratanalert S, Zhang K, Zhang F, Yan H, Chiu W, Bathe M 2016 Science 352 1534
 Google Scholar
Google Scholar
[47] Liu W, Zhong H, Wang R, Seeman N C 2011 Angew. Chem. Int. Ed. 50 264
 Google Scholar
Google Scholar
[48] Iinuma R, Ke Y, Jungmann R, Schlichthaerle T, Woehrstein J B, Yin P 2014 Science 344 65
 Google Scholar
Google Scholar
[49] Wagenbauer K F, Sigl C, Dietz H 2017 Nature 552 78
 Google Scholar
Google Scholar
[50] Tikhomirov G, Petersen P, Qian L 2017 Nature 552 67
 Google Scholar
Google Scholar
[51] Simmel F C, Yurke B, Singh H R 2019 Chem. Rev. 119 6326
[52] Douglas S M, Bachelet I, Church G M 2012 Science 335 831
 Google Scholar
Google Scholar
[53] Zhu D, Pei H, Yao G, Wang L, Su S, Chao J, Wang L, Aldalbahi A, Song S, Shi J, Hu J, Fan C, Zuo X 2016 Adv. Mater. 28 6860
 Google Scholar
Google Scholar
[54] Lund K, Manzo A J, Dabby N, Michelotti N, Johnson-Buck A, Nangreave J, Taylor S, Pei R, Stojanovic M N, Walter N G, Winfree E, Yan H 2010 Nature 465 206
 Google Scholar
Google Scholar
[55] Wickham S F J, Endo M, Katsuda Y, Hidaka K, Bath J, Sugiyama H, Turberfield A J 2011 Nat. Nanotechnol. 6 166
 Google Scholar
Google Scholar
[56] Steinhauer C, Jungmann R, Sobey T L, Simmel F C, Tinnefeld P 2009 Angew. Chem. Int. Ed. 48 8870
 Google Scholar
Google Scholar
[57] Li J, Dai J, Jiang S, Xie M, Zhai T, Guo L, Cao S, Xing S, Qu Z, Zhao Y, Wang F, Yang Y, Liu L, Zuo X, Wang L, Yan H, Fan C 2020 Nat. Commun. 11 2185
 Google Scholar
Google Scholar
[58] Lu N, Pei H, Ge Z, Simmons C R, Yan H, Fan C 2012 J. Am. Chem. Soc. 134 13148
 Google Scholar
Google Scholar
[59] Yang Y, Wang J, Shigematsu H, Xu W, Shih W M, Rothman J E, Lin C 2016 Nat. Chem. 8 476
 Google Scholar
Google Scholar
[60] Zheng J, Constantinou P E, Micheel C, Alivisatos A P, Kiehl R A, Seeman N C 2006 Nano Lett. 6 1502
 Google Scholar
Google Scholar
[61] Sharma J, Chhabra R, Cheng A, Brownell J, Liu Y, Yan H 2009 Science 323 112
 Google Scholar
Google Scholar
[62] Lo P K, Karam P, Aldaye F A, McLaughlin C K, Hamblin G D, Cosa G, Sleiman H F 2010 Nat. Chem. 2 319
 Google Scholar
Google Scholar
[63] Liu W, Halverson J, Tian Y, Tkachenko A V, Gang O 2016 Nat. Chem. 8 867
 Google Scholar
Google Scholar
[64] Tian Y, Lhermitte J R, Bai L, Vo T, Xin H L, Li H, Li R, Fukuto M, Yager K G, Kahn J S, Xiong Y, Minevich B, Kumar S K, Gang O 2020 Nat. Mater. 19 789
 Google Scholar
Google Scholar
[65] Yao G, Li J, Li Q, Chen X, Liu X, Wang F, Qu Z, Ge Z, Narayanan R P, Williams D, Pei H, Zuo X, Wang L, Yan H, Feringa B L, Fan C 2020 Nat. Mater. 19 781
 Google Scholar
Google Scholar
[66] Liu J, Geng Y, Pound E, Gyawali S, Ashton J R, Hickey J, Woolley A T, Harb J N 2011 ACS Nano 5 2240
 Google Scholar
Google Scholar
[67] Li N, Shang Y, Xu R, Jiang Q, Liu J, Wang L, Cheng Z, Ding B 2019 J. Am. Chem. Soc. 141 17968
 Google Scholar
Google Scholar
[68] Sun W, Boulais E, Hakobyan Y, Wang W L, Guan A, Bathe M, Yin P 2014 Science 346 1258361
 Google Scholar
Google Scholar
[69] Pal S, Varghese R, Deng Z, Zhao Z, Kumar A, Yan H, Liu Y 2011 Angew. Chem. Int. Ed. 50 4176
 Google Scholar
Google Scholar
[70] Jia S, Wang J, Xie M, Sun J, Liu H, Zhang Y, Chao J, Li J, Wang L, Lin J, Gothelf K V, Fan C 2019 Nat. Commun. 10 5597
 Google Scholar
Google Scholar
[71] Liu X, Zhang F, Jing X, Pan M, Liu P, Li W, Zhu B, Li J, Chen H, Wang L, Lin J, Liu Y, Zhao D, Yan H, Fan C 2018 Nature 559 593
 Google Scholar
Google Scholar
[72] Shang Y, Li N, Liu S, Wang L, Wang Z G, Zhang Z, Ding B 2020 Adv. Mater. 32 2000294
 Google Scholar
Google Scholar
[73] 贾思思, 晁洁, 樊春海, 柳华杰 2014 化学进展 26 695
Jia S, Chao J, Fan C, Liu H 2014 Prog. Chem. 26 695
[74] Voigt N V, Tørring T, Rotaru A, Jacobsen M F, Ravnsbæk J B, Subramani R, Mamdouh W, Kjems J, Mokhir A, Besenbacher F, Gothelf K V 2010 Nat. Nanotechnol. 5 200
 Google Scholar
Google Scholar
[75] Liu H, Tørring T, Dong M, Rosen C B, Besenbacher F, Gothelf K V 2010 J. Am. Chem. Soc. 132 18054
 Google Scholar
Google Scholar
[76] Tokura Y, Harvey S, Chen C, Wu Y, Ng D Y W, Weil T 2018 Angew. Chem. Int. Ed. 57 1587
 Google Scholar
Google Scholar
[77] Winterwerber P, Harvey S, Ng D Y W, Weil T 2020 Angew. Chem. Int. Ed. 59 6144
 Google Scholar
Google Scholar
[78] Ke G, Liu M, Jiang S, Qi X, Yang Y R, Wootten S, Zhang F, Zhu Z, Liu Y, Yang C J, Yan H 2016 Angew. Chem. Int. Ed. 55 7483
 Google Scholar
Google Scholar
[79] Fu Y, Zeng D, Chao J, Jin Y, Zhang Z, Liu H, Li D, Ma H, Huang Q, Gothelf K V, Fan C 2013 J. Am. Chem. Soc. 135 696
 Google Scholar
Google Scholar
[80] Xin L, Zhou C, Yang Z, Liu D 2013 Small 9 3088
 Google Scholar
Google Scholar
[81] Hansen M H, Blakskjaer P, Petersen L K, Hansen T H, Hojfeldt J W, Gothelf K V, Hansen N J V 2009 J. Am. Chem. Soc. 131 1322
 Google Scholar
Google Scholar
[82] Wilner O I, Weizmann Y, Gill R, Lioubashevski O, Freeman R, Willner I 2009 Nat. Nanotechnol. 4 249
 Google Scholar
Google Scholar
[83] Fu J, Liu M, Liu Y, Woodbury N W, Yan H 2012 J. Am. Chem. Soc. 134 5516
 Google Scholar
Google Scholar
[84] Ke Y, Lindsay S, Chang Y, Liu Y, Yan H 2008 Science 319 180
 Google Scholar
Google Scholar
[85] Subramanian H K K, Chakraborty B, Sha R, Seeman N C 2011 Nano Lett. 11 910
 Google Scholar
Google Scholar
[86] Zhang H, Chao J, Pan D, Liu H, Qiang Y, Liu K, Cui C, Chen J, Huang Q, Hu J, Wang L, Huang W, Shi Y, Fan C 2017 Nat. Commun. 8 14738
 Google Scholar
Google Scholar
[87] Pei H, Lu N, Wen Y, Song S, Liu Y, Yan H, Fan C 2010 Adv. Mater. 22 4754
 Google Scholar
Google Scholar
[88] Kühler P, Roller E M, Schreiber R, Liedl T, Lohmüller T, Feldmann J 2014 Nano Lett. 14 2914
 Google Scholar
Google Scholar
[89] Fang W, Jia S, Chao J, Wang L, Duan X, Liu H, Li Q, Zuo X, Wang L, Wang L, Liu N, Fan C 2019 Sci. Adv. 5 eaau4506
 Google Scholar
Google Scholar
[90] Li Z, Zhao B, Wang D, Wen Y, Liu G, Dong H, Song S, Fan C 2014 ACS Appl. Mater. Interfaces 6 17944
 Google Scholar
Google Scholar
[91] 张祎男, 王丽华, 柳华杰, 樊春海 2017 物理学报 66 147101
 Google Scholar
Google Scholar
Zhang Y, Wang L, Liu H, Fan C 2017 Acta Phys. Sin. 66 147101
 Google Scholar
Google Scholar
[92] Simoncelli S, Roller E M, Urban P, Schreiber R, Turberfield A J, Liedl T, Lohmüller T 2016 ACS Nano 10 9809
 Google Scholar
Google Scholar
[93] Gu H, Chao J, Xiao S J, Seeman N C 2010 Nature 465 202
 Google Scholar
Google Scholar
[94] Thubagere A J, Li W, Johnson R F, Chen Z, Doroudi S, Lee Y L, Izatt G, Wittman S, Srinivas N, Woods D, Winfree E, Qian L 2017 Science 357 eaan6558
 Google Scholar
Google Scholar
[95] Kopperger E, List J, Madhira S, Rothfischer F, Lamb D C, Simmel F C 2018 Science 359 296
 Google Scholar
Google Scholar
[96] Lauback S, Mattioli K R, Marras A E, Armstrong M, Rudibaugh T P, Sooryakumar R, Castro C E 2018 Nat. Commun. 9 1446
 Google Scholar
Google Scholar
[97] Li S, Jiang Q, Liu S, Zhang Y, Tian Y, Song C, Wang J, Zou Y, Anderson G J, Han J Y, Chang Y, Liu Y, Zhang C, Chen L, Zhou G, Nie G, Yan H, Ding B, Zhao Y 2018 Nat. Biotechnol. 36 258
 Google Scholar
Google Scholar
[98] Wiraja C, Zhu Y, Lio D C S, Yeo D C, Xie M, Fang W, Li Q, Zheng M, Van Steensel M, Wang L, Fan C, Xu C 2019 Nat. Commun. 10 1147
 Google Scholar
Google Scholar
[99] Ijäs H, Hakaste I, Shen B, Kostiainen M A, Linko V 2019 ACS Nano 13 5959
 Google Scholar
Google Scholar
[100] Burns J R, Lamarre B, Pyne A L B, Noble J E, Ryadnov M G 2018 ACS Synth. Biol. 7 767
 Google Scholar
Google Scholar
[101] Chen Q, Liu H, Lee W, Sun Y, Zhu D, Pei H, Fan C, Fan X 2013 Lab Chip 13 3351
 Google Scholar
Google Scholar
[102] Jungmann R, Avendaño M S, Woehrstein J B, Dai M, Shih W M, Yin P 2014 Nat. Methods 11 313
 Google Scholar
Google Scholar
[103] Vogele K, List J, Pardatscher G, Holland N B, Simmel F C, Pirzer T 2016 ACS Nano 10 11377
 Google Scholar
Google Scholar
[104] Kuzyk A, Schreiber R, Fan Z, Pardatscher G, Roller E M, Högele A, Simmel F C, Govorov A O, Liedl T 2012 Nature 483 311
 Google Scholar
Google Scholar
[105] Man T, Ji W, Liu X, Zhang C, Li L, Pei H, Fan C 2019 ACS Nano 13 4826
 Google Scholar
Google Scholar
[106] Zhou C, Duan X, Liu N 2015 Nat. Commun. 6 8102
 Google Scholar
Google Scholar
[107] Klein W P, Schmidt C N, Rapp B, Takabayashi S, Knowlton W B, Lee J, Yurke B, Hughes W L, Graugnard E, Kuang W 2013 Nano Lett. 13 3850
 Google Scholar
Google Scholar
[108] Gür F N, McPolin C P T, Raza S, Mayer M, Roth D J, Steiner A M, Löffler M, Fery A, Brongersma M L, Zayats A V, König T A F, Schmidt T L 2018 Nano Lett. 18 7323
 Google Scholar
Google Scholar
[109] Shen X, Asenjo-Garcia A, Liu Q, Jiang Q, García de Abajo F J, Liu N, Ding B 2013 Nano Lett. 13 2128
 Google Scholar
Google Scholar
[110] Lan X, Lu X, Shen C, Ke Y, Ni W, Wang Q 2015 J. Am. Chem. Soc. 137 457
 Google Scholar
Google Scholar
[111] Dai X, Li Q, Aldalbahi A, Wang L, Fan C, Liu X 2020 Nano Lett. 20 5604
 Google Scholar
Google Scholar
[112] Deng Z X, Mao C D 2004 Angew. Chem. Int. Ed. 43 4068
 Google Scholar
Google Scholar
[113] Becerril H A, Woolley A T 2007 Small 3 1534
 Google Scholar
Google Scholar
[114] Diagne C T, Brun C, Gasparutto D, Baillin X, Tiron R 2016 ACS Nano 10 6458
 Google Scholar
Google Scholar
[115] Knudsen J B, Liu L, Bank Kodal A L, Madsen M, Li Q, Song J, Woehrstein J B, Wickham S F J, Strauss M T, Schueder F, Vinther J, Krissanaprasit A, Gudnason D, Smith A A A, Ogaki R, Zelikin A N, Besenbacher F, Birkedal V, Yin P, Shih W M, Jungmann R, Dong M, Gothelf K V 2015 Nat. Nanotechnol. 10 892
 Google Scholar
Google Scholar
[116] Tapio K, Leppiniemi J, Shen B, Hytönen V P, Fritzsche W, Toppari J J 2016 Nano Lett. 16 6780
 Google Scholar
Google Scholar
[117] Sun W, Shen J, Zhao Z, Arellano N, Rettner C, Tang J, Cao T, Zhou Z, Ta T, Streit J K, Fagan J A, Schaus T, Zheng M, Han S J, Shih W M, Maune H T, Yin P 2020 Science 368 874
 Google Scholar
Google Scholar
[118] Zhao M, Chen Y, Wang K, Zhang Z, Streit J K, Fagan J A, Tang J, Zheng M, Yang C, Zhu Z, Sun W 2020 Science 368 878
 Google Scholar
Google Scholar
[119] Zhang Y, Mao X, Li F, Li M, Jing X, Ge Z, Wang L, Liu K, Zhang H, Fan C, Zuo X 2020 Angew. Chem. Int. Ed. 59 4892
 Google Scholar
Google Scholar
[120] Adleman L 1994 Science 266 1021
 Google Scholar
Google Scholar
[121] Bai M, Chen F, Cao X, Zhao Y, Xue J, Yu X, Fan C, Zhao Y 2020 Angew. Chem. Int. Ed. 59 13267
 Google Scholar
Google Scholar
[122] Liu H, Wang J, Song S, Fan C, Gothelf K V 2015 Nat. Commun. 6 10089
 Google Scholar
Google Scholar
[123] Chao J, Wang J, Wang F, Ouyang X, Kopperger E, Liu H, Li Q, Shi J, Wang L, Hu J, Wang L, Huang W, Simmel F C, Fan C 2019 Nat. Mater. 18 273
 Google Scholar
Google Scholar
[124] Zhang Y, Wang F, Chao J, Xie M, Liu H, Pan M, Kopperger E, Liu X, Li Q, Shi J, Wang L, Hu J, Wang L, Simmel F C, Fan C 2019 Nat. Commun. 10 5469
 Google Scholar
Google Scholar
[125] Fan S, Wang D, Cheng J, Liu Y, Luo T, Cui D, Ke Y, Song J 2020 Angew. Chem. Int. Ed. 59 12991
 Google Scholar
Google Scholar
[126] Wickham S F J, Bath J, Katsuda Y, Endo M, Hidaka K, Sugiyama H, Turberfield A J 2012 Nat. Nanotechnol. 7 169
 Google Scholar
Google Scholar
[127] Clelland C T, Risca V, Bancroft C 1999 Nature 399 533
 Google Scholar
Google Scholar
[128] Song J, Li Z, Wang P, Meyer T, Mao C, Ke Y 2017 Science 357 eaan3377
 Google Scholar
Google Scholar
-
图 1 典型框架核酸结构: DNA瓦块和DNA折纸结构 (a) DNA瓦块组装成的二维晶格[28]; (b) DNA四面体[20]; (c) 类富勒烯结构[21]; (d) DNA折纸设计图及几种二维平面折纸结构[22]; (e) 球形[40]、花鸟图案[42]和兔子[41]线框DNA折纸结构; (f) DNA纳米花瓶结构[43]; (g) 16个折纸模块组成的蒙娜丽莎图案[50]
Fig. 1. Typical FNAs: DNA tile and DNA origami: (a) DNA four-way junction[28]; (b) DNA tetrahedron[20]; (c) DNA buckyball self-assembled by three-point-star DNA tiles[21]; (d) 2D DNA origami structures[22]; (e) sphere[40], flower-and-bird pattern[42] and bunny-shape[41] wireframe DNA origami structures; (f) nanoflask DNA origami structure with complex curvatures[43]; (g) a Mona Lisa pattern self-assembled by 16 DNA origami tiles[50].
图 2 框架核酸介导纳米颗粒组装 (a) DNA瓦块介导AuNPs组装成二维阵列[60]; (b) 三角形DNA纳米管封装的AuNP线[62]; (c) DNA折纸模块介导AuNPs形成平面阵列[63]; (d) DNA单链编码的AuNPs组装成分支状类分子结构[65]
Fig. 2. FNAs-directed nanoparticles assembly: (a) 2D AuNP arrays self-assembled by DNA tiles [60]; (b) AuNP lines size-selective encapsulated within triangular DNA nanotubes[62]; (c) 2D AuNP arrays directed by DNA origami tiles[63]; (d) branched molecule-like structures self-assembled by single-stranded DNA encoded AuNPs[65].
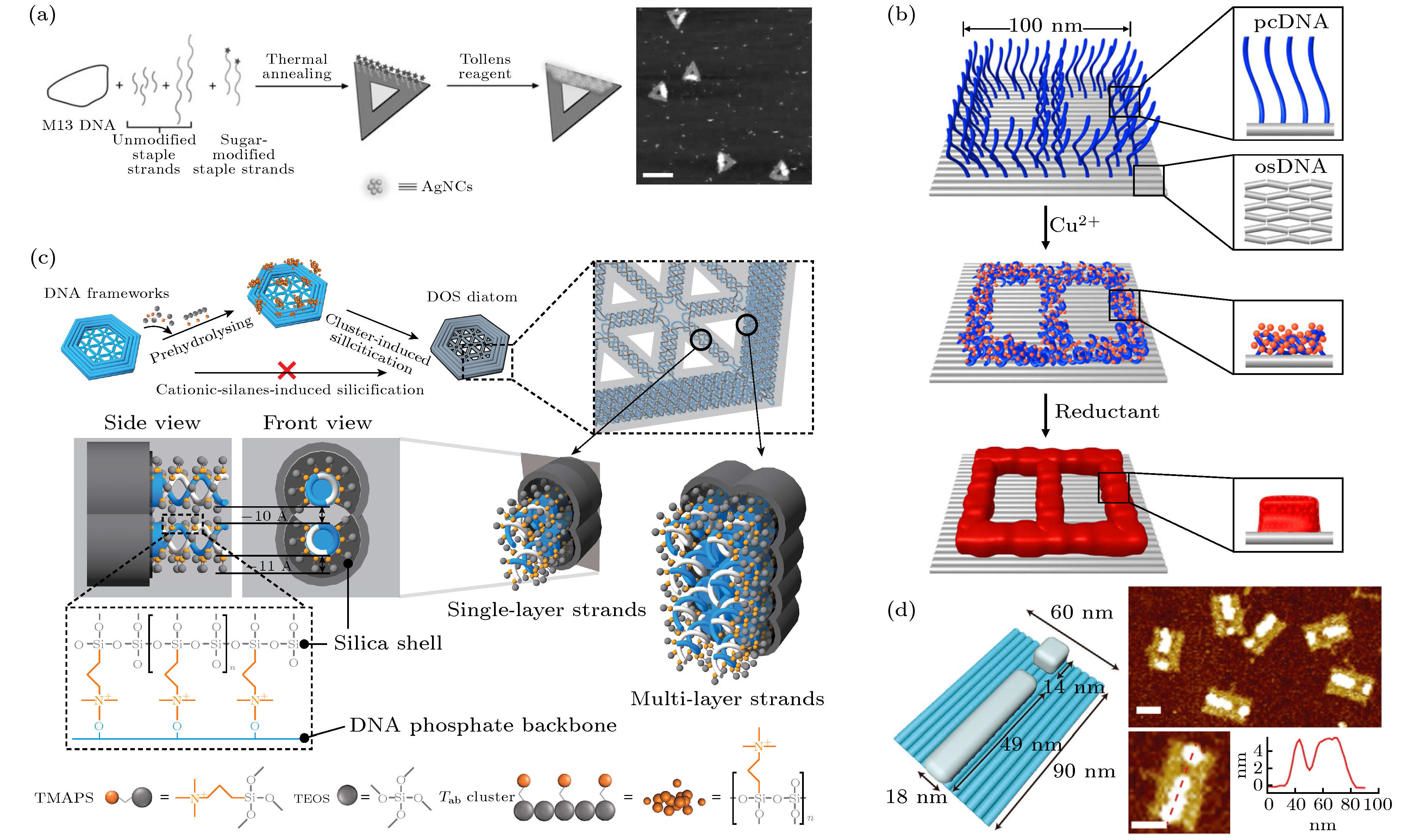
图 3 框架核酸介导原位生长 (a) 三角形DNA折纸上定点金属化形成银纳米簇[69]; (b) DNA折纸上选择性金属化构建8字形图案[70]; (c) DNA-二氧化硅复合材料的制备[71]; (d) DNA折纸上定点合成“i”形二氧化硅纳米结构[72]
Fig. 3. FNAs-directed in-situ growth of nanomaterials: (a) Silver nanoclusters synthesized on DNA origami[69]; (b) selective DNA condensation and metallization on DNA origami for fabricating a digit 8 pattern[70]; (c) DNA origami silicification diatom-mimicking structures[71]; (d) site-specific synthesis of “i-pattern” silica nanostructure on DNA origami[72].
图 4 框架核酸构建单分子反应器 (a) DNA 折纸上单分子化学键断裂反应[74]; (b) DNA折纸上光诱导多巴胺聚合反应[77]; (c) DNA折纸上酶通路调控系统[78]; (d) DNA纳米管中GOx和HRP的酶级联反应[79]; (e) DNA机器可逆调控酶级联反应[80]
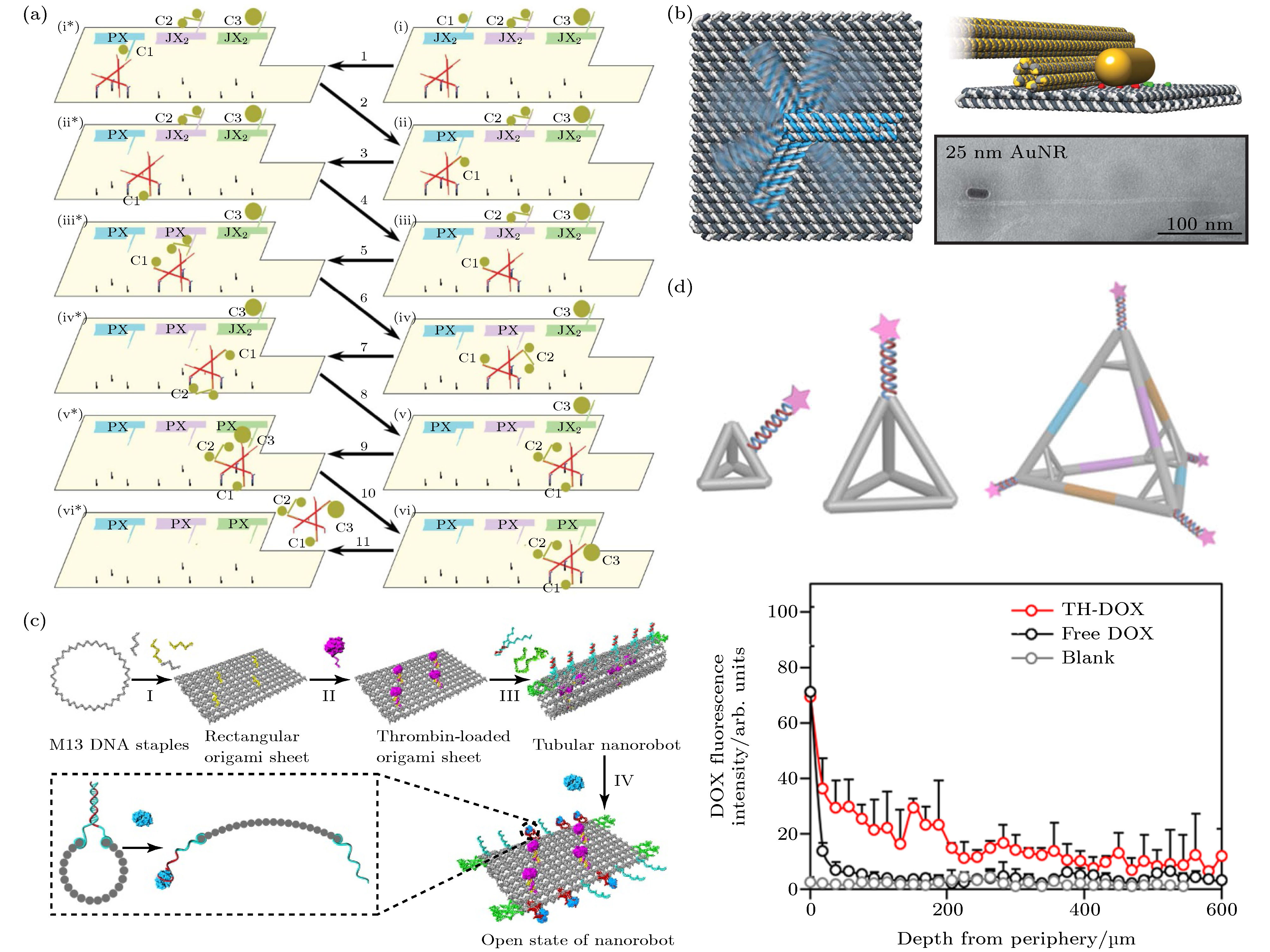
Fig. 4. FNAs used for single molecule reactors: (a) Single-molecule chemical cleavage reactions on DNA origami[74]; (b) light-triggered polydopamine formation on DNA origami[77]; (c) enzyme pathway regulation system on a rectangular DNA origami platform[78]; (d) bienzyme cascade of GOx and HRP in a DNA origami nanotube[79]; (e) reversible regulation of enzyme cascade reaction by a DNA machine[80].
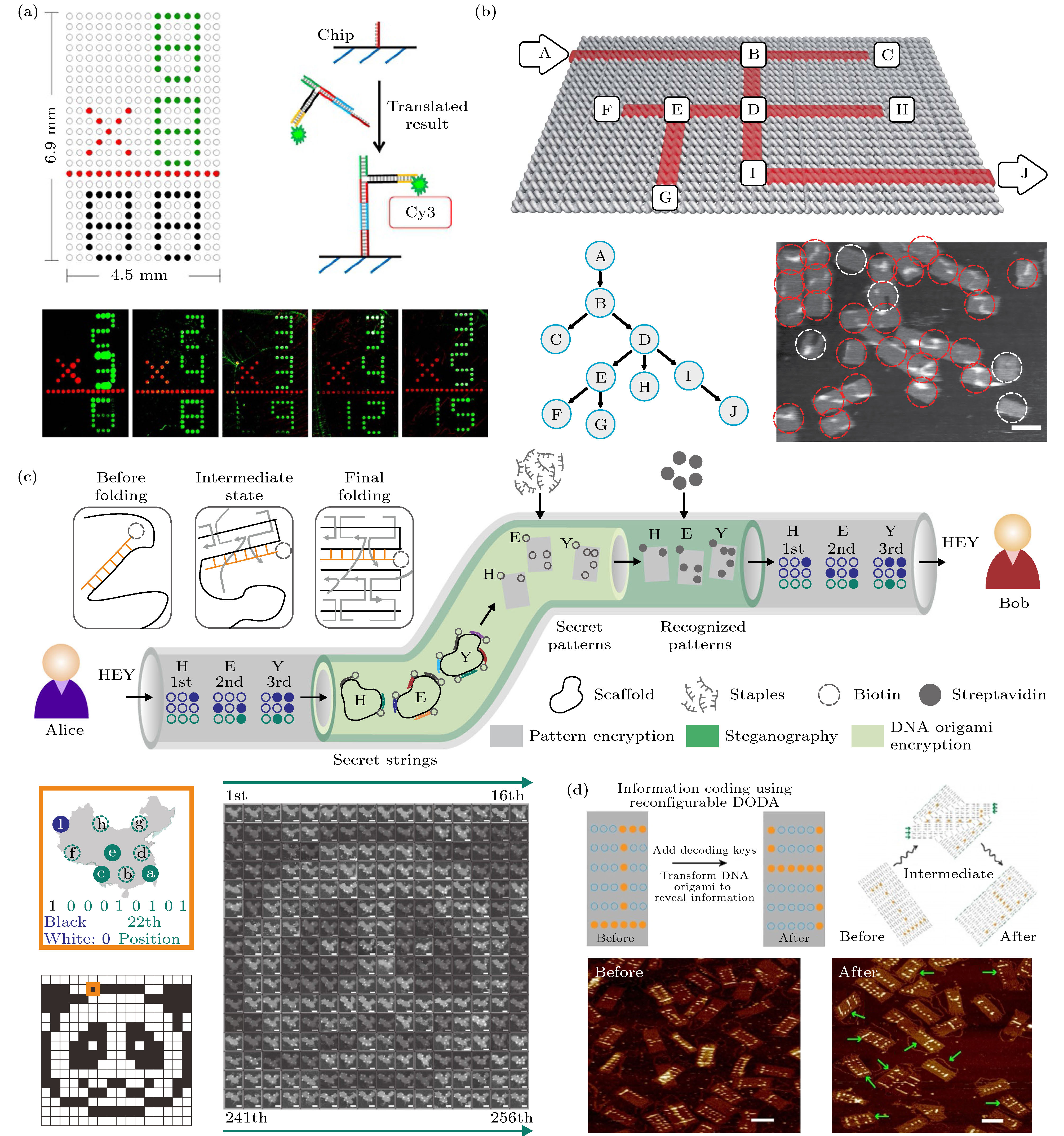
图 5 框架核酸构建单分子传感器 (a) DNA 折纸上设计的V形探针检测目标RNA[84]; (b) 字母图案的DNA折纸用于SNP检测[85]; (c) DNA四面体探针检测目标DNA[87]; (d) AuNP二聚体检测染料分子的SERS信号[88]; (e) AuNP四聚体对SERS信号的单分子水平定点、定量检测[89]
Fig. 5. FNAs used for single molecule sensing: (a) Detection of the target RNA by hybridization with V-shaped probe stretched from a DNA origami[84]; (b) SNP detection with alphabetic patterned origami structures [85]; (c) recognition of the target DNA with a DNA tetrahedral structured probe [87]; (d) DNA origami-templated AuNP dimers for SERS[88]; (e) DNA origami-templated tetrameric Au nanoclusters for quantizing single-molecule SERS[89].
图 6 框架核酸用于分子装载和输运 (a) DNA 折纸上的分子装配线[93]; (b) 电场驱动DNA纳米机械臂旋转并使金纳米棒运动[95]; (c) 凝血酶功能化的DNA纳米机器人[97]; (d) 3种用于经皮给药的DNA四面体结构[98]
Fig. 6. FNAs used for cargos loading and transporting: (a) Molecular assembly line on DNA origami[93]; (b) electrically actuated rotation of a nanorobotic arm, moving a gold nanorod[95]; (c) DNA origami nanocapsule actuated by changing pH[97]; (d) 3DNA tetrahedrons for transdermal drug delivery[98].
图 7 框架核酸的纳米光学应用 (a) 基于DNA四面体的光流体激光器实验装置[101]; (b) DNA-PAINT[102]; (c) DNA折纸上线性排列的AuNPs产生光波导[103]; (d) AuNPs在DNA折纸上的左旋和右旋排列[104]; (e) 四面体DNA折纸组装的金纳米棒手性超分子[105]; (f) 金纳米棒在DNA折纸上的动态行走[106]
Fig. 7. FNAs used for nanophotonics: (a) Optofluidic lasers based on a DNA tetrahedron[101]; (b) DNA-PAINT [102]; (c) waveguide on the line of AuNPs arranged by a DNA origami[103]; (d) AuNP helices on DNA origami[104]; (e) tetrahedral DNA origami-templated plasmonic metamolecules[105]; (f) Au nanorod walking on DNA origami[106].
图 8 框架核酸构建纳电子器件 (a) DNA折纸到SiO2基底的直接图案转移[114]; (b) 聚合物线在DNA折纸上形成的“U”形图案[115]; (c) DNA瓦块组装AuNP构建单电子晶体管[116]; (d) DNA折纸模板制备高度致密的CNT平行阵列[117]
Fig. 8. FNAs used for nanoelectronics: (a) Pattern transferring from DNA origami into SiO2[114]; (b) polymer binding to the DNA origami with a “U” shaped pattern[113]; (c) DNA tile-templated single electron nanoelectronics[116]; (d) CNT alignment based on trench-like DNA templates[117].
图 9 框架核酸构建信息处理器件 (a) DNA 折纸分子计算器[122]; (b) DNA单分子巡航机器人解迷宫[123]; (c) DNA折纸加密系统[124]; (d) DNA折纸多米诺阵列编码信息[125]
Fig. 9. FNAs used for information processing: (a) DNA origami calculator[122]; (b) single-molecule DNA navigator for solving maze on the 2 D origami[123]; (c) DNA origami cryptography system[124]; (d) DNA origami domino array for coding information[125].
-
[1] Chang C Z, Zhang J, Feng X, et al. 2013 Science 340 167
 Google Scholar
Google Scholar
[2] Yu R, Zhang W, Zhang H J, Zhang S C, Dai X, Fang Z 2010 Science 329 61
 Google Scholar
Google Scholar
[3] Li Y 2018 Phys. Rev. A 98 012336
 Google Scholar
Google Scholar
[4] Fei H, Dong J, Arellano-Jiménez M J, Ye G, Dong Kim N, Samuel E L G, Peng Z, Zhu Z, Qin F, Bao J, Yacaman M J, Ajayan P M, Chen D, Tour J M 2015 Nat. Commun. 6 8668
 Google Scholar
Google Scholar
[5] Qiao B, Wang A, Yang X, Allard L F, Jiang Z, Cui Y, Liu J, Li J, Zhang T 2011 Nat. Chem. 3 634
 Google Scholar
Google Scholar
[6] Eigler D M, Schweizer E K 1990 Nature 344 524
 Google Scholar
Google Scholar
[7] Crommie M F, Lutz C P, Eigler D M 1993 Science 262 218
 Google Scholar
Google Scholar
[8] Hänsel W, Hommelhoff P, Hänsch T W, Reichel J 2001 Nature 413 498
 Google Scholar
Google Scholar
[9] Feynman R P 1960 Eng. Sci. 23 22
[10] Suess B, Weigand J E 2008 RNA Biol. 5 24
 Google Scholar
Google Scholar
[11] Maurel M C, Leclerc F, Hervé G 2020 Chem. Rev. 120 4898
 Google Scholar
Google Scholar
[12] Chen L L, Yang L 2015 RNA Biol. 12 381
 Google Scholar
Google Scholar
[13] Parkinson G N, Lee M P H, Neidle S 2002 Nature 417 876
 Google Scholar
Google Scholar
[14] Day H A, Pavlou P, Waller Z A E 2014 Bioorg. Med. Chem. 22 4407
 Google Scholar
Google Scholar
[15] Jones M R, Seeman N C, Mirkin C A 2015 Science 347 1260901
 Google Scholar
Google Scholar
[16] 樊春海, 刘冬生 2011 DNA纳米技术: 分子传感、计算与机器 (北京: 科学出版社) 第19页
Fan C, Liu D 2011 DNA Nanotechnology: Moelcular Sensoring, Computation and Machines. (Beijing: Science Press) p19 (in Chinese)
[17] Winfree E, Liu F, Wenzler L A, Seeman N C 1998 Nature 394 539
 Google Scholar
Google Scholar
[18] Yan H, Park S H, Finkelstein G, Reif J H, LaBean T H 2003 Science 301 1882
 Google Scholar
Google Scholar
[19] Lin C, Liu Y, Rinker S, Yan H 2006 ChemPhysChem 7 1641
 Google Scholar
Google Scholar
[20] Goodman R P, Schaap I A T, Tardin C F, Erben C M, Berry R M, Schmidt C F, Turberfield A J 2005 Science 310 1661
 Google Scholar
Google Scholar
[21] He Y, Ye T, Su M, Zhang C, Ribbe A E, Jiang W, Mao C 2008 Nature 452 198
 Google Scholar
Google Scholar
[22] Rothemund P W K 2006 Nature 440 297
 Google Scholar
Google Scholar
[23] Ge Z, Gu H, Li Q, Fan C 2018 J. Am. Chem. Soc. 140 17808
 Google Scholar
Google Scholar
[24] Wang F, Zhang X, Liu X, Fan C, Li Q 2019 Small 15 1900013
 Google Scholar
Google Scholar
[25] Song X, Reif J 2019 ACS Nano 13 6256
 Google Scholar
Google Scholar
[26] Yang F, Li Q, Wang L, Zhang G J, Fan C 2018 ACS Sens. 3 903
 Google Scholar
Google Scholar
[27] Hu Q, Li H, Wang L, Gu H, Fan C 2019 Chem. Rev. 119 6459
[28] Seeman N C 1982 J. Theor. Biol. 99 237
 Google Scholar
Google Scholar
[29] Kuzuya A, Wang R, Sha R, Seeman N C 2007 Nano Lett. 7 1757
 Google Scholar
Google Scholar
[30] Ding B, Sha R, Seeman N C 2004 J. Am. Chem. Soc. 126 10230
 Google Scholar
Google Scholar
[31] He Y, Chen Y, Liu H, Ribbe A E, Mao C 2005 J. Am. Chem. Soc. 127 12202
 Google Scholar
Google Scholar
[32] Chen J, Seeman N C 1991 Nature 350 631
 Google Scholar
Google Scholar
[33] Zheng J, Birktoft J J, Chen Y, Wang T, Sha R, Constantinou P E, Ginell S L, Mao C, Seeman N C 2009 Nature 461 74
 Google Scholar
Google Scholar
[34] Guo P 2010 Nat. Nanotechnol. 5 833
 Google Scholar
Google Scholar
[35] Afonin K A, Bindewald E, Yaghoubian A J, Voss N, Jacovetty E, Shapiro B A, Jaeger L 2010 Nat. Nanotechnol. 5 676
 Google Scholar
Google Scholar
[36] Severcan I, Geary C, Chworos A, Voss N, Jacovetty E, Jaeger L 2010 Nat. Chem. 2 772
 Google Scholar
Google Scholar
[37] 钱璐璐, 汪颖, 张钊, 赵健, 潘敦, 张益, 刘强, 樊春海, 胡钧, 贺林 2006 科学通报 51 2860
 Google Scholar
Google Scholar
Qian L, Wang Y, Zhang Z, Zhao J, Pan D, Zhang Y, Liu Q, Fan C, Hu J, He L 2006 Chin. Sci. Bull. 51 2860
 Google Scholar
Google Scholar
[38] Andersen E S, Dong M, Nielsen M M, Jahn K, Subramani R, Mamdouh W, Golas M M, Sander B, Stark H, Oliveira C L P, Pedersen J S, Birkedal V, Besenbacher F, Gothelf K V, Kjems J 2009 Nature 459 73
 Google Scholar
Google Scholar
[39] Douglas S M, Dietz H, Liedl T, Högberg B, Graf F, Shih W M 2009 Nature 459 414
 Google Scholar
Google Scholar
[40] Han D, Pal S, Yang Y, Jiang S, Nangreave J, Liu Y, Yan H 2013 Science 339 1412
 Google Scholar
Google Scholar
[41] Benson E, Mohammed A, Gardell J, Masich S, Czeizler E, Orponen P, Högberg B 2015 Nature 523 441
 Google Scholar
Google Scholar
[42] Zhang F, Jiang S, Wu S, Li Y, Mao C, Liu Y, Yan H 2015 Nat. Nanotechnol. 10 779
 Google Scholar
Google Scholar
[43] Han D, Pal S, Nangreave J, Deng Z, Liu Y, Yan H 2011 Science 332 342
 Google Scholar
Google Scholar
[44] Williams S, Lund K, Lin C, Wonka P, Lindsay S, Yan H 2009 LNCS 5347 90
[45] Douglas S M, Marblestone A H, Teerapittayanon S, Vazquez A, Church G M, Shih W M 2009 Nucl. Acids Res. 37 5001
 Google Scholar
Google Scholar
[46] Veneziano R, Ratanalert S, Zhang K, Zhang F, Yan H, Chiu W, Bathe M 2016 Science 352 1534
 Google Scholar
Google Scholar
[47] Liu W, Zhong H, Wang R, Seeman N C 2011 Angew. Chem. Int. Ed. 50 264
 Google Scholar
Google Scholar
[48] Iinuma R, Ke Y, Jungmann R, Schlichthaerle T, Woehrstein J B, Yin P 2014 Science 344 65
 Google Scholar
Google Scholar
[49] Wagenbauer K F, Sigl C, Dietz H 2017 Nature 552 78
 Google Scholar
Google Scholar
[50] Tikhomirov G, Petersen P, Qian L 2017 Nature 552 67
 Google Scholar
Google Scholar
[51] Simmel F C, Yurke B, Singh H R 2019 Chem. Rev. 119 6326
[52] Douglas S M, Bachelet I, Church G M 2012 Science 335 831
 Google Scholar
Google Scholar
[53] Zhu D, Pei H, Yao G, Wang L, Su S, Chao J, Wang L, Aldalbahi A, Song S, Shi J, Hu J, Fan C, Zuo X 2016 Adv. Mater. 28 6860
 Google Scholar
Google Scholar
[54] Lund K, Manzo A J, Dabby N, Michelotti N, Johnson-Buck A, Nangreave J, Taylor S, Pei R, Stojanovic M N, Walter N G, Winfree E, Yan H 2010 Nature 465 206
 Google Scholar
Google Scholar
[55] Wickham S F J, Endo M, Katsuda Y, Hidaka K, Bath J, Sugiyama H, Turberfield A J 2011 Nat. Nanotechnol. 6 166
 Google Scholar
Google Scholar
[56] Steinhauer C, Jungmann R, Sobey T L, Simmel F C, Tinnefeld P 2009 Angew. Chem. Int. Ed. 48 8870
 Google Scholar
Google Scholar
[57] Li J, Dai J, Jiang S, Xie M, Zhai T, Guo L, Cao S, Xing S, Qu Z, Zhao Y, Wang F, Yang Y, Liu L, Zuo X, Wang L, Yan H, Fan C 2020 Nat. Commun. 11 2185
 Google Scholar
Google Scholar
[58] Lu N, Pei H, Ge Z, Simmons C R, Yan H, Fan C 2012 J. Am. Chem. Soc. 134 13148
 Google Scholar
Google Scholar
[59] Yang Y, Wang J, Shigematsu H, Xu W, Shih W M, Rothman J E, Lin C 2016 Nat. Chem. 8 476
 Google Scholar
Google Scholar
[60] Zheng J, Constantinou P E, Micheel C, Alivisatos A P, Kiehl R A, Seeman N C 2006 Nano Lett. 6 1502
 Google Scholar
Google Scholar
[61] Sharma J, Chhabra R, Cheng A, Brownell J, Liu Y, Yan H 2009 Science 323 112
 Google Scholar
Google Scholar
[62] Lo P K, Karam P, Aldaye F A, McLaughlin C K, Hamblin G D, Cosa G, Sleiman H F 2010 Nat. Chem. 2 319
 Google Scholar
Google Scholar
[63] Liu W, Halverson J, Tian Y, Tkachenko A V, Gang O 2016 Nat. Chem. 8 867
 Google Scholar
Google Scholar
[64] Tian Y, Lhermitte J R, Bai L, Vo T, Xin H L, Li H, Li R, Fukuto M, Yager K G, Kahn J S, Xiong Y, Minevich B, Kumar S K, Gang O 2020 Nat. Mater. 19 789
 Google Scholar
Google Scholar
[65] Yao G, Li J, Li Q, Chen X, Liu X, Wang F, Qu Z, Ge Z, Narayanan R P, Williams D, Pei H, Zuo X, Wang L, Yan H, Feringa B L, Fan C 2020 Nat. Mater. 19 781
 Google Scholar
Google Scholar
[66] Liu J, Geng Y, Pound E, Gyawali S, Ashton J R, Hickey J, Woolley A T, Harb J N 2011 ACS Nano 5 2240
 Google Scholar
Google Scholar
[67] Li N, Shang Y, Xu R, Jiang Q, Liu J, Wang L, Cheng Z, Ding B 2019 J. Am. Chem. Soc. 141 17968
 Google Scholar
Google Scholar
[68] Sun W, Boulais E, Hakobyan Y, Wang W L, Guan A, Bathe M, Yin P 2014 Science 346 1258361
 Google Scholar
Google Scholar
[69] Pal S, Varghese R, Deng Z, Zhao Z, Kumar A, Yan H, Liu Y 2011 Angew. Chem. Int. Ed. 50 4176
 Google Scholar
Google Scholar
[70] Jia S, Wang J, Xie M, Sun J, Liu H, Zhang Y, Chao J, Li J, Wang L, Lin J, Gothelf K V, Fan C 2019 Nat. Commun. 10 5597
 Google Scholar
Google Scholar
[71] Liu X, Zhang F, Jing X, Pan M, Liu P, Li W, Zhu B, Li J, Chen H, Wang L, Lin J, Liu Y, Zhao D, Yan H, Fan C 2018 Nature 559 593
 Google Scholar
Google Scholar
[72] Shang Y, Li N, Liu S, Wang L, Wang Z G, Zhang Z, Ding B 2020 Adv. Mater. 32 2000294
 Google Scholar
Google Scholar
[73] 贾思思, 晁洁, 樊春海, 柳华杰 2014 化学进展 26 695
Jia S, Chao J, Fan C, Liu H 2014 Prog. Chem. 26 695
[74] Voigt N V, Tørring T, Rotaru A, Jacobsen M F, Ravnsbæk J B, Subramani R, Mamdouh W, Kjems J, Mokhir A, Besenbacher F, Gothelf K V 2010 Nat. Nanotechnol. 5 200
 Google Scholar
Google Scholar
[75] Liu H, Tørring T, Dong M, Rosen C B, Besenbacher F, Gothelf K V 2010 J. Am. Chem. Soc. 132 18054
 Google Scholar
Google Scholar
[76] Tokura Y, Harvey S, Chen C, Wu Y, Ng D Y W, Weil T 2018 Angew. Chem. Int. Ed. 57 1587
 Google Scholar
Google Scholar
[77] Winterwerber P, Harvey S, Ng D Y W, Weil T 2020 Angew. Chem. Int. Ed. 59 6144
 Google Scholar
Google Scholar
[78] Ke G, Liu M, Jiang S, Qi X, Yang Y R, Wootten S, Zhang F, Zhu Z, Liu Y, Yang C J, Yan H 2016 Angew. Chem. Int. Ed. 55 7483
 Google Scholar
Google Scholar
[79] Fu Y, Zeng D, Chao J, Jin Y, Zhang Z, Liu H, Li D, Ma H, Huang Q, Gothelf K V, Fan C 2013 J. Am. Chem. Soc. 135 696
 Google Scholar
Google Scholar
[80] Xin L, Zhou C, Yang Z, Liu D 2013 Small 9 3088
 Google Scholar
Google Scholar
[81] Hansen M H, Blakskjaer P, Petersen L K, Hansen T H, Hojfeldt J W, Gothelf K V, Hansen N J V 2009 J. Am. Chem. Soc. 131 1322
 Google Scholar
Google Scholar
[82] Wilner O I, Weizmann Y, Gill R, Lioubashevski O, Freeman R, Willner I 2009 Nat. Nanotechnol. 4 249
 Google Scholar
Google Scholar
[83] Fu J, Liu M, Liu Y, Woodbury N W, Yan H 2012 J. Am. Chem. Soc. 134 5516
 Google Scholar
Google Scholar
[84] Ke Y, Lindsay S, Chang Y, Liu Y, Yan H 2008 Science 319 180
 Google Scholar
Google Scholar
[85] Subramanian H K K, Chakraborty B, Sha R, Seeman N C 2011 Nano Lett. 11 910
 Google Scholar
Google Scholar
[86] Zhang H, Chao J, Pan D, Liu H, Qiang Y, Liu K, Cui C, Chen J, Huang Q, Hu J, Wang L, Huang W, Shi Y, Fan C 2017 Nat. Commun. 8 14738
 Google Scholar
Google Scholar
[87] Pei H, Lu N, Wen Y, Song S, Liu Y, Yan H, Fan C 2010 Adv. Mater. 22 4754
 Google Scholar
Google Scholar
[88] Kühler P, Roller E M, Schreiber R, Liedl T, Lohmüller T, Feldmann J 2014 Nano Lett. 14 2914
 Google Scholar
Google Scholar
[89] Fang W, Jia S, Chao J, Wang L, Duan X, Liu H, Li Q, Zuo X, Wang L, Wang L, Liu N, Fan C 2019 Sci. Adv. 5 eaau4506
 Google Scholar
Google Scholar
[90] Li Z, Zhao B, Wang D, Wen Y, Liu G, Dong H, Song S, Fan C 2014 ACS Appl. Mater. Interfaces 6 17944
 Google Scholar
Google Scholar
[91] 张祎男, 王丽华, 柳华杰, 樊春海 2017 物理学报 66 147101
 Google Scholar
Google Scholar
Zhang Y, Wang L, Liu H, Fan C 2017 Acta Phys. Sin. 66 147101
 Google Scholar
Google Scholar
[92] Simoncelli S, Roller E M, Urban P, Schreiber R, Turberfield A J, Liedl T, Lohmüller T 2016 ACS Nano 10 9809
 Google Scholar
Google Scholar
[93] Gu H, Chao J, Xiao S J, Seeman N C 2010 Nature 465 202
 Google Scholar
Google Scholar
[94] Thubagere A J, Li W, Johnson R F, Chen Z, Doroudi S, Lee Y L, Izatt G, Wittman S, Srinivas N, Woods D, Winfree E, Qian L 2017 Science 357 eaan6558
 Google Scholar
Google Scholar
[95] Kopperger E, List J, Madhira S, Rothfischer F, Lamb D C, Simmel F C 2018 Science 359 296
 Google Scholar
Google Scholar
[96] Lauback S, Mattioli K R, Marras A E, Armstrong M, Rudibaugh T P, Sooryakumar R, Castro C E 2018 Nat. Commun. 9 1446
 Google Scholar
Google Scholar
[97] Li S, Jiang Q, Liu S, Zhang Y, Tian Y, Song C, Wang J, Zou Y, Anderson G J, Han J Y, Chang Y, Liu Y, Zhang C, Chen L, Zhou G, Nie G, Yan H, Ding B, Zhao Y 2018 Nat. Biotechnol. 36 258
 Google Scholar
Google Scholar
[98] Wiraja C, Zhu Y, Lio D C S, Yeo D C, Xie M, Fang W, Li Q, Zheng M, Van Steensel M, Wang L, Fan C, Xu C 2019 Nat. Commun. 10 1147
 Google Scholar
Google Scholar
[99] Ijäs H, Hakaste I, Shen B, Kostiainen M A, Linko V 2019 ACS Nano 13 5959
 Google Scholar
Google Scholar
[100] Burns J R, Lamarre B, Pyne A L B, Noble J E, Ryadnov M G 2018 ACS Synth. Biol. 7 767
 Google Scholar
Google Scholar
[101] Chen Q, Liu H, Lee W, Sun Y, Zhu D, Pei H, Fan C, Fan X 2013 Lab Chip 13 3351
 Google Scholar
Google Scholar
[102] Jungmann R, Avendaño M S, Woehrstein J B, Dai M, Shih W M, Yin P 2014 Nat. Methods 11 313
 Google Scholar
Google Scholar
[103] Vogele K, List J, Pardatscher G, Holland N B, Simmel F C, Pirzer T 2016 ACS Nano 10 11377
 Google Scholar
Google Scholar
[104] Kuzyk A, Schreiber R, Fan Z, Pardatscher G, Roller E M, Högele A, Simmel F C, Govorov A O, Liedl T 2012 Nature 483 311
 Google Scholar
Google Scholar
[105] Man T, Ji W, Liu X, Zhang C, Li L, Pei H, Fan C 2019 ACS Nano 13 4826
 Google Scholar
Google Scholar
[106] Zhou C, Duan X, Liu N 2015 Nat. Commun. 6 8102
 Google Scholar
Google Scholar
[107] Klein W P, Schmidt C N, Rapp B, Takabayashi S, Knowlton W B, Lee J, Yurke B, Hughes W L, Graugnard E, Kuang W 2013 Nano Lett. 13 3850
 Google Scholar
Google Scholar
[108] Gür F N, McPolin C P T, Raza S, Mayer M, Roth D J, Steiner A M, Löffler M, Fery A, Brongersma M L, Zayats A V, König T A F, Schmidt T L 2018 Nano Lett. 18 7323
 Google Scholar
Google Scholar
[109] Shen X, Asenjo-Garcia A, Liu Q, Jiang Q, García de Abajo F J, Liu N, Ding B 2013 Nano Lett. 13 2128
 Google Scholar
Google Scholar
[110] Lan X, Lu X, Shen C, Ke Y, Ni W, Wang Q 2015 J. Am. Chem. Soc. 137 457
 Google Scholar
Google Scholar
[111] Dai X, Li Q, Aldalbahi A, Wang L, Fan C, Liu X 2020 Nano Lett. 20 5604
 Google Scholar
Google Scholar
[112] Deng Z X, Mao C D 2004 Angew. Chem. Int. Ed. 43 4068
 Google Scholar
Google Scholar
[113] Becerril H A, Woolley A T 2007 Small 3 1534
 Google Scholar
Google Scholar
[114] Diagne C T, Brun C, Gasparutto D, Baillin X, Tiron R 2016 ACS Nano 10 6458
 Google Scholar
Google Scholar
[115] Knudsen J B, Liu L, Bank Kodal A L, Madsen M, Li Q, Song J, Woehrstein J B, Wickham S F J, Strauss M T, Schueder F, Vinther J, Krissanaprasit A, Gudnason D, Smith A A A, Ogaki R, Zelikin A N, Besenbacher F, Birkedal V, Yin P, Shih W M, Jungmann R, Dong M, Gothelf K V 2015 Nat. Nanotechnol. 10 892
 Google Scholar
Google Scholar
[116] Tapio K, Leppiniemi J, Shen B, Hytönen V P, Fritzsche W, Toppari J J 2016 Nano Lett. 16 6780
 Google Scholar
Google Scholar
[117] Sun W, Shen J, Zhao Z, Arellano N, Rettner C, Tang J, Cao T, Zhou Z, Ta T, Streit J K, Fagan J A, Schaus T, Zheng M, Han S J, Shih W M, Maune H T, Yin P 2020 Science 368 874
 Google Scholar
Google Scholar
[118] Zhao M, Chen Y, Wang K, Zhang Z, Streit J K, Fagan J A, Tang J, Zheng M, Yang C, Zhu Z, Sun W 2020 Science 368 878
 Google Scholar
Google Scholar
[119] Zhang Y, Mao X, Li F, Li M, Jing X, Ge Z, Wang L, Liu K, Zhang H, Fan C, Zuo X 2020 Angew. Chem. Int. Ed. 59 4892
 Google Scholar
Google Scholar
[120] Adleman L 1994 Science 266 1021
 Google Scholar
Google Scholar
[121] Bai M, Chen F, Cao X, Zhao Y, Xue J, Yu X, Fan C, Zhao Y 2020 Angew. Chem. Int. Ed. 59 13267
 Google Scholar
Google Scholar
[122] Liu H, Wang J, Song S, Fan C, Gothelf K V 2015 Nat. Commun. 6 10089
 Google Scholar
Google Scholar
[123] Chao J, Wang J, Wang F, Ouyang X, Kopperger E, Liu H, Li Q, Shi J, Wang L, Hu J, Wang L, Huang W, Simmel F C, Fan C 2019 Nat. Mater. 18 273
 Google Scholar
Google Scholar
[124] Zhang Y, Wang F, Chao J, Xie M, Liu H, Pan M, Kopperger E, Liu X, Li Q, Shi J, Wang L, Hu J, Wang L, Simmel F C, Fan C 2019 Nat. Commun. 10 5469
 Google Scholar
Google Scholar
[125] Fan S, Wang D, Cheng J, Liu Y, Luo T, Cui D, Ke Y, Song J 2020 Angew. Chem. Int. Ed. 59 12991
 Google Scholar
Google Scholar
[126] Wickham S F J, Bath J, Katsuda Y, Endo M, Hidaka K, Sugiyama H, Turberfield A J 2012 Nat. Nanotechnol. 7 169
 Google Scholar
Google Scholar
[127] Clelland C T, Risca V, Bancroft C 1999 Nature 399 533
 Google Scholar
Google Scholar
[128] Song J, Li Z, Wang P, Meyer T, Mao C, Ke Y 2017 Science 357 eaan3377
 Google Scholar
Google Scholar
计量
- 文章访问数: 17334
- PDF下载量: 380
- 被引次数: 0













 下载:
下载: