-
病毒是一种在活细胞内寄生并以复制方式增殖的非细胞型生物. 不同病毒侵入细胞的方式不同, 但大都需要通过结合至细胞表面特定的受体蛋白或脂质结构来实现细胞内化, 从而启动入侵程序和感染宿主细胞. 因此揭示病毒结合和内化侵入细胞的具体过程及机制有助于从源头上开发靶向药物或疫苗. 本文以流感病毒和冠状病毒为例, 首先介绍了流感病毒的结构, 其与细胞膜上特定种类蛋白或脂质结构的结合方式, 其完成细胞内吞的途径及诱导其内化的细胞因子种类, 以及侵入细胞后的作用过程, 继而对冠状病毒尤其针对目前仍在全球范围内肆意传播的新型冠状病毒(SARS-CoV-2), 扼要阐述了其结构特点、与细胞受体血管紧张素转化酶ACE2的结合及实现细胞内化的研究进展.Viruses are acellular organisms that must be parasitized in living cells and proliferated by replication. Although different viruses invade cells in different ways, they mainly initiate the invasion process through binding to specific receptor proteins or lipid structures on the cell surface for the following cellular internalization. Thus revealing the interaction process and underlying mechanism between viruses and cell membranes will be helpful in developing targeted drugs or vaccines from the source. In this review, the influenza virus and coronavirus are taken for example. We will first discuss the structure of influenza viruses, their binding modes with cell membranes, the way of realizing cell endocytosis and the cytokines involved in this process. After that, recent research progress of coronavirus especially the novel coronavirus SARS-CoV-2, including its structural characteristics, its binding with cell receptor ACE2 and the following cellular internalization, is briefly introduced.
-
Keywords:
- influenza virus /
- novel coronavirus SARS-CoV-2 /
- cellular internalization /
- clathrin-mediated endocytosis
[1] Sieczkarski S B, Whittaker G R 2002 J. Gen. Virol. 83 1535
 Google Scholar
Google Scholar
[2] Nabi I R, Le P U 2003 J. Cell Biol. 161 673
 Google Scholar
Google Scholar
[3] Seto W H, Tsang D, Yung R W H, Ching T Y, Ng T K, Ho M, Ho L M, Peiris J S M, Auth A E S G H 2003 Lancet 361 1519
 Google Scholar
Google Scholar
[4] Wenzel R P, Edmond M B 2009 N. Engl. J. Med. 361 1991
 Google Scholar
Google Scholar
[5] de Groot R J, Baker S C, Baric R S, Brown C S, Drosten C, Enjuanes L, Fouchier R A M, Galiano M, Gorbalenya A E, Memish Z A, Perlman S, Poon L L M, Snijder E J, Stephens G M, Woo P C Y, Zaki A M, Zambon M, Ziebuhr J 2013 J. Virol. 87 7790
 Google Scholar
Google Scholar
[6] Gao R B, Cao B, Hu Y W, et al. 2013 N. Engl. J. Med. 368 1888
 Google Scholar
Google Scholar
[7] Song F X, Shi N N, Shan F, Zhang Z Y, Shen J, Lu H Z, Ling Y, Jiang Y B, Shi Y X 2020 Radiology 295 210
 Google Scholar
Google Scholar
[8] Li M, Ding H M, Lin M H, Yin F F, Song L, Mao X H, Li F, Ge Z L, Wang L H, Zuo X L, Ma Y Q, Fan C H 2019 J. Am. Chem. Soc. 141 18910
 Google Scholar
Google Scholar
[9] Wang H, Jiang C 2009 Sci. China. C Life Sci. 52 464
 Google Scholar
Google Scholar
[10] Lakadamyali M, Rust M J, Babcock H P, Zhuang X 2003 Proc. Natl. Acad. Sci. U. S. A. 100 9280
 Google Scholar
Google Scholar
[11] Fontana J, Steven A C 2013 J. Virol. 87 5621
 Google Scholar
Google Scholar
[12] Liu S L, Zhang Z L, Tian Z Q, Zhao H S, Liu H, Sun E Z, Xiao G F, Zhang W, Wang H Z, Pang D W 2011 ACS Nano 6 141
 Google Scholar
Google Scholar
[13] Babcock H P, Chen C, Zhuang X W 2004 Biophys. J. 87 2749
 Google Scholar
Google Scholar
[14] Wang M, Cao R, Zhang L, Yang X, Liu J, Xu M, Shi Z, Hu Z, Zhong W, Xiao G 2020 Cell Res. 30 269
 Google Scholar
Google Scholar
[15] Lu H 2020 Biosci. Trends 14 69
 Google Scholar
Google Scholar
[16] Li R F, Hou Y L, Huang J C, Pan W Q, Ma Q H, Shi Y X, Li C F, Zhao J, Jia Z H, Jiang H M, Zheng K, Huang S X, Dai J, Li X B, Hou X T, Wang L, Zhong N A, Yang Z F 2020 Pharmacol. Res. 156 104761
 Google Scholar
Google Scholar
[17] Dadonaite B, Vijayakrishnan S, Fodor E, Bhella D, Hutchinson E C 2016 J. Gen. Virol. 97 1755
 Google Scholar
Google Scholar
[18] Mosley V M, Wyckoff R W G 1946 Nature 157 263
 Google Scholar
Google Scholar
[19] Taylor A R, Sharp D G, Beard D, Beard J W, Dingle J H, Feller A E 1943 J. Immunol. 47 261
 Google Scholar
Google Scholar
[20] Sharp D G, Taylor A R, McLean I W, Jr Beard D, Beard J W, Fellew A E, Dingle J H 1943 Science 98 307
 Google Scholar
Google Scholar
[21] Choppin P W, Murphy J S, Tamm I 1960 J. Exp. Med. 112 945
 Google Scholar
Google Scholar
[22] Kilbourne E D, Murphy J S 1960 J. Exp. Med. 111 387
 Google Scholar
Google Scholar
[23] Kumlin U, Olofsson S, Dimock K, Arnberg N 2008 Influenza Other Resp. 2 147
 Google Scholar
Google Scholar
[24] Rogers G N, D'Souza B L 1989 Virology 173 317
 Google Scholar
Google Scholar
[25] Connor R J, Kawaoka Y, Webster R G, Paulson J C 1994 Virology 205 17
 Google Scholar
Google Scholar
[26] Peng W J, de Vries R P, Grant O C, Thompson A J, McBride R, Tsogtbaatar B, Lee P S, Razi N, Wilson I A, Woods R J, Paulson J C 2017 Cell Host Microbe 21 23
 Google Scholar
Google Scholar
[27] Ng W C, Liong S, Tate M D, Irimura T, Denda-Nagai K, Brooks A G, Londrigan S L, Reading P C 2014 J. Virol. 88 1659
 Google Scholar
Google Scholar
[28] Goronzy I N, Rawle R J, Boxer S G, Kasson P M 2018 Chem. Sci. 9 2340
 Google Scholar
Google Scholar
[29] Ding H M, Li J, Chen N, Hu X J, Yang X F, Guo L J, Li Q, Zuo X L, Wang L H, Ma Y Q, Fan C H 2018 Acs Central Sci 4 1344
 Google Scholar
Google Scholar
[30] Verma D K, Gupta D, Lal S K 2018 Viruses 10 650
 Google Scholar
Google Scholar
[31] Byrd-Leotis L, Jia N, Dutta S, Trost J F, Gao C, Cummings S F, Braulke T, Muller-Loennies S, Heimburg-Molinaro J, Steinhauer D A, Cummings R D 2019 Sci. Adv. 5 eaav2554
 Google Scholar
Google Scholar
[32] Ng W C, Londrigan S L, Nasr N, Cunningham A L, Turville S, Brooks A G, Reading P C 2016 J. Virol. 90 206
 Google Scholar
Google Scholar
[33] Oh N, Park J H 2014 Int. J. Nanomed. 9 51
 Google Scholar
Google Scholar
[34] Geiser M 2010 J. Aerosol Med. Pulm. Drug Deliv. 23 207
 Google Scholar
Google Scholar
[35] Dourmashkin R R, Tyrrell D A 1974 J. Gen. Virol. 24 129
 Google Scholar
Google Scholar
[36] Matlin K S, Reggio H, Helenius A, Simons K 1981 J. Cell Biol. 91 601
 Google Scholar
Google Scholar
[37] Conner S D, Schmid S L 2003 Nature 422 37
 Google Scholar
Google Scholar
[38] Nichols B J, Lippincott-Schwartz J 2001 Trends Cell Biol. 11 406
 Google Scholar
Google Scholar
[39] Li Y, Yue T T, Yang K, Zhang X R 2012 Biomaterials 33 4965
 Google Scholar
Google Scholar
[40] Rust M J, Lakadamyali M, Zhang F, Zhuang X W 2004 Nat. Struct. Mol. Biol. 11 567
 Google Scholar
Google Scholar
[41] Sieczkarski S B, Whittaker G R 2002 J. Virol. 76 10455
 Google Scholar
Google Scholar
[42] Sun E Z, Liu A A, Zhang Z L, Liu S L, Tian Z Q, Pang D W 2017 ACS Nano 11 4395
 Google Scholar
Google Scholar
[43] Thomsen P, Roepstorff K, Stahlhut M, van Deurs B, Riezman H 2002 Mol. Biol. Cell 13 238
 Google Scholar
Google Scholar
[44] Schnitzer J E, Oh P, Pinney E, Allard J 1994 J. Cell Biol. 127 1217
 Google Scholar
Google Scholar
[45] Orlandi P A, Fishman P H 1998 J. Cell Biol. 141 905
 Google Scholar
Google Scholar
[46] Nunes-Correia I, Eulalio A, Nir S, De Lima M C P 2004 Cell. Mol. Biol. Lett. 9 47
[47] Mercer J, Helenius A 2009 Nat. Cell Biol. 11 510
 Google Scholar
Google Scholar
[48] de Vries E, Tscherne D M, Wienholts M J, Cobos-Jimenez V, Scholte F, Garcia-Sastre A, Rottier P J, de Haan C A 2011 PLoS Pathog. 7 e1001329
 Google Scholar
Google Scholar
[49] Rossman J S, Leser G P, Lamb R A 2012 J. Virol. 86 10950
 Google Scholar
Google Scholar
[50] Carroll S M, Paulson J C 1985 Virus Res. 3 165
 Google Scholar
Google Scholar
[51] Williams S P, Robertson J S 1993 Virology 196 660
 Google Scholar
Google Scholar
[52] de Vries E, de Vries R P, Wienholts M J, Floris C E, Jacobs M S, van den Heuvel A, Rottier P J, de Haan C A 2012 Proc. Natl. Acad. Sci. U. S. A. 109 7457
 Google Scholar
Google Scholar
[53] Conner S D, Schmid S L 2003 J. Cell Biol. 162 773
 Google Scholar
Google Scholar
[54] Wang G, Jiang L, Wang J, Zhang J, Kong F, Li Q, Yan Y, Huang S, Zhao Y, Liang L, Li J, Sun N, Hu Y, Shi W, Deng G, Chen P, Liu L, Zeng X, Tian G, Bu Z, Chen H, Li C 2020 J. Virol. 94
 Google Scholar
Google Scholar
[55] Chen C, Zhuang X W 2008 Proc. Natl. Acad. Sci. U.S.A. 105 11790
 Google Scholar
Google Scholar
[56] Tayyari F, Marchant D, Moraes T J, Duan W M, Mastrangelo P, Hegele R G 2011 Nat. Med. 17 1132
 Google Scholar
Google Scholar
[57] Melen K, Tynell J, Fagerlund R, Roussel P, Hernandez-Verdun D, Julkunen I 2012 Virol J. 9 167
 Google Scholar
Google Scholar
[58] Murayama R, Harada Y, Shibata T, Kuroda K, Hayakawa S, Shimizu K, Tanaka T 2007 Biochem. Biophys. Res. Commun. 362 880
 Google Scholar
Google Scholar
[59] Chan C M, Chu H, Zhang A J, Leung L H, Sze K H, Kao R Y, Chik K K, To K K, Chan J F, Chen H, Jin D Y, Liu L, Yuen K Y 2016 Virology 494 78
 Google Scholar
Google Scholar
[60] Ji Q J, Yuan B, Lu X M, Yang K, Ma Y Q 2016 Small 12 1140
 Google Scholar
Google Scholar
[61] Geijtenbeek T B H, Gringhuis S I 2009 Nat. Rev. Immunol. 9 465
 Google Scholar
Google Scholar
[62] van Kooyk Y 2008 Biochem. Soc. Trans. 36 1478
 Google Scholar
Google Scholar
[63] Robinson M J, Sancho D, Slack E C, LeibundGut-Landmann S, Sousa C R E 2006 Nat. Immunol. 7 1258
 Google Scholar
Google Scholar
[64] Drickamer K 1992 Nature 360 183
 Google Scholar
Google Scholar
[65] Reading P C, Miller J L, Anders E M 2000 J. Virol. 74 5190
 Google Scholar
Google Scholar
[66] Valladeau J, Ravel O, Dezutter-Dambuyant C, Moore K, Kleijmeer M, Liu Y, Duvert-Frances V, Vincent C, Schmitt D, Davoust J, Caux C, Lebecque S, Saeland S 2000 Immunity 12 71
 Google Scholar
Google Scholar
[67] Cohen G B, Ren R B, Baltimore D 1995 Cell 80 237
 Google Scholar
Google Scholar
[68] Fujioka Y, Tsuda M, Nanbo A, Hattori T, Sasaki J, Sasaki T, Miyazaki T, Ohba Y 2013 Nat Commun 4 2763
 Google Scholar
Google Scholar
[69] Fujioka Y, Nishide S, Ose T, et al. 2018 Cell Host Microbe 23 809
 Google Scholar
Google Scholar
[70] Ehrhardt C, Marjuki H, Wolff T, Nuernberg B, Planz O, Pleschka S, Ludwig S 2006 Cell. Microbiol. 8 1336
 Google Scholar
Google Scholar
[71] Zhu L, Ly H, Liang Y 2014 J. Virol. 88 417
 Google Scholar
Google Scholar
[72] Chu V C, Whittaker G R 2004 Proc. Natl. Acad. Sci. U. S. A. 101 18153
 Google Scholar
Google Scholar
[73] Lu X, Xu P P, Ding H M, Yu Y S, Huo D, Ma Y Q 2019 Nat Commun 10 4520
 Google Scholar
Google Scholar
[74] Hamilton B S, Whittaker G R, Daniel S 2012 Viruses 4 1144
 Google Scholar
Google Scholar
[75] Fodor E 2013 Acta Virol. 57 113
 Google Scholar
Google Scholar
[76] Akarsu H, Burmeister W P, Petosa C, Petit I, Muller C W, Ruigrok R W H, Baudin F 2003 EMBO J. 22 4646
 Google Scholar
Google Scholar
[77] Dou D, Revol R, Ostbye H, Wang H, Daniels R 2018 Front Immunol 9 1581
 Google Scholar
Google Scholar
[78] Zhang J, Pekosz A, Lamb R A 2000 J. Virol. 74 4634
 Google Scholar
Google Scholar
[79] Dahmani I, Ludwig K, Chiantia S 2019 Biosci. Rep. 39 16
 Google Scholar
Google Scholar
[80] Pillay T S 2020 J. Clin. Pathol. 73 366
 Google Scholar
Google Scholar
[81] Han Q, Lin Q, Jin S, You L 2020 J. Infect. 80 373
 Google Scholar
Google Scholar
[82] Zhu N, Zhang D, Wang W, Li X, Yang B, Song J, Zhao X, Huang B, Shi W, Lu R, Niu P, Zhan F, Ma X, Wang D, Xu W, Wu G, Gao G F, Tan W, China Novel Coronavirus I, Research T 2020 N. Engl. J. Med. 382 727
 Google Scholar
Google Scholar
[83] Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, Wang W, Song H, Huang B, Zhu N, Bi Y, Ma X, Zhan F, Wang L, Hu T, Zhou H, Hu Z, Zhou W, Zhao L, Chen J, Meng Y, Wang J, Lin Y, Yuan J, Xie Z, Ma J, Liu W J, Wang D, Xu W, Holmes E C, Gao G F, Wu G, Chen W, Shi W, Tan W 2020 Lancet 395 565
 Google Scholar
Google Scholar
[84] Hoffmann M, Kleine-Weber H, Schroeder S, Kruger N, Herrler T, Erichsen S, Schiergens T S, Herrler G, Wu N H, Nitsche A, Muller M A, Drosten C, Pohlmann S 2020 Cell 181 271
 Google Scholar
Google Scholar
[85] Wang Q, Qiu Y, Li J Y, Zhou Z J, Liao C H, Ge X Y 2020 Virol. Sin. 35 337
 Google Scholar
Google Scholar
[86] Cheng Z, Zhou J, To K K, Chu H, Li C, Wang D, Yang D, Zheng S, Hao K, Bosse Y, Obeidat M, Brandsma C A, Song Y Q, Chen Y, Zheng B J, Li L, Yuen K Y 2015 J. Infect. Dis. 212 1214
 Google Scholar
Google Scholar
[87] Offringa A, Montijn R, Singh S, Paul M, Pinto Y M, Pinto-Sietsma S J 2020 Eur Heart J Cardiovasc Pharmacother pvaa053 doi: 10.1093/ehjcvp/pvaa053
[88] Sohail A, Nutini A 2020 Prog. Biophys. Mol. Biol. 155 29
 Google Scholar
Google Scholar
[89] Ng M L, Tan S H, See E E, Ooi E E, Ling A E 2003 J. Med. Virol. 71 323
 Google Scholar
Google Scholar
[90] Yang Z Y, Huang Y, Ganesh L, Leung K, Kong W P, Schwartz O, Subbarao K, Nabel G J 2004 J. Virol. 78 5642
 Google Scholar
Google Scholar
[91] Huang I C, Bosch B J, Li F, Li W H, Lee K H, Ghiran S, Vasilieva N, Dermody T S, Harrison S C, Dormitzer P R, Farzan M, Rottier P J M, Choe H 2006 J. Biol. Chem. 281 3198
 Google Scholar
Google Scholar
[92] Inoue Y, Tanaka N, Tanaka Y, Inoue S, Morita K, Zhuang M, Hattori T, Sugamura K 2007 J. Virol. 81 8722
 Google Scholar
Google Scholar
[93] Wang H L, Yang P, Liu K T, Guo F, Zhang Y L, Zhang G Y, Jiang C Y 2008 Cell Res. 18 290
 Google Scholar
Google Scholar
[94] Hofmann H, Pohlmann S 2004 Trends Microbiol. 12 466
 Google Scholar
Google Scholar
-
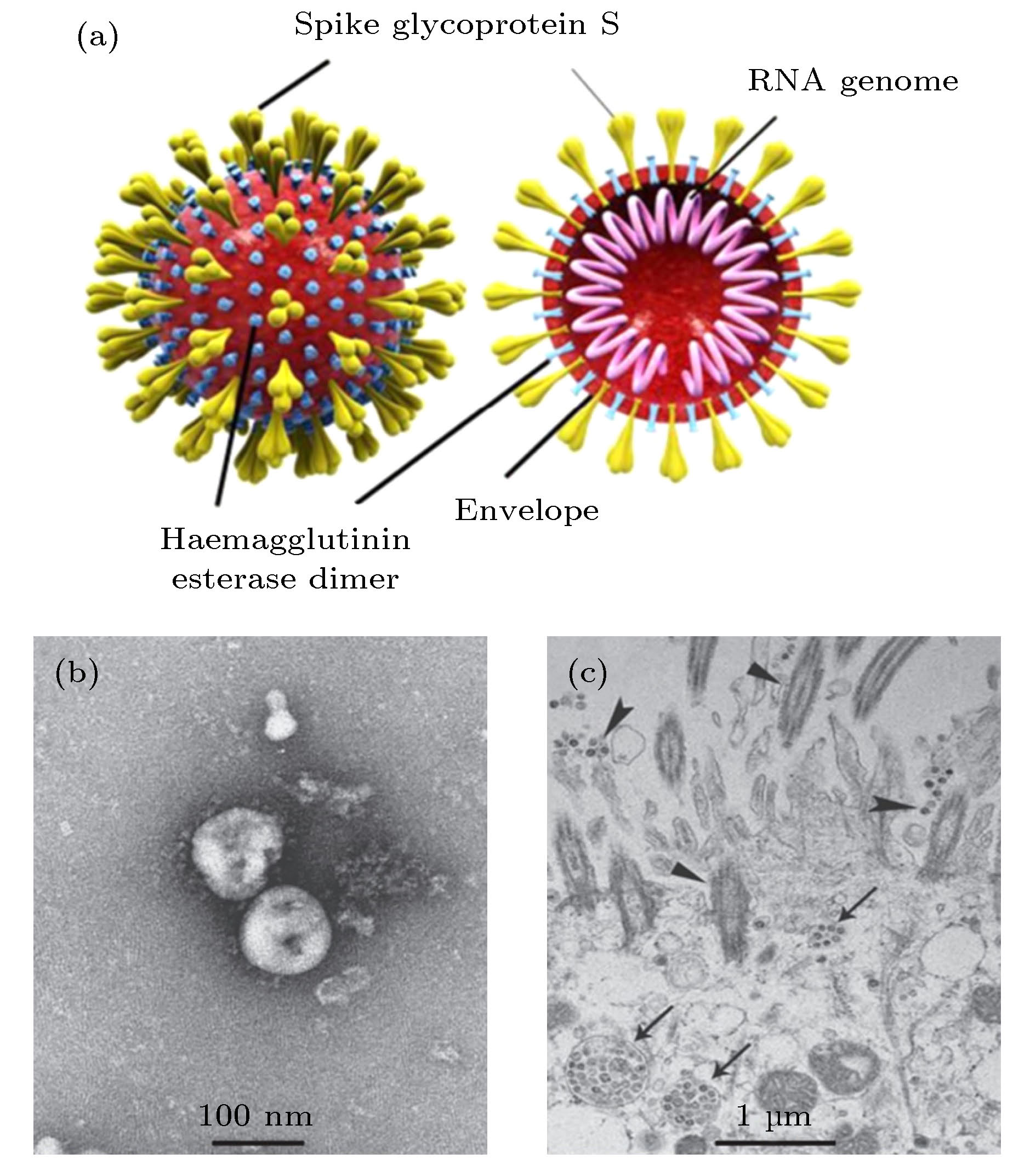
图 1 流感病毒的结构与形状[17,21] (a) 丝状、杆状与球状流感病毒模型图, 流感病毒是包被病毒, 最外层是脂质膜, 膜上含有HA和NA两种糖蛋白及M2通道蛋白, 膜下是基质蛋白M1, 丝状病毒的基因组位于远端, 球状病毒的基因组位于中心; (b) 丝状病毒和(c) 球状病毒的电子显微镜照片
Fig. 1. Composition and shape of influenza virus[17,21]: (a) Filamentous, rod-shaped and globular influenza virus models; (b), (c) representative electron microscopy images of filamentous and globular viruses, respectively.
图 2 流感病毒在细胞表面结合及内化示意图. 流感病毒与细胞表面的糖蛋白或糖脂末端的唾液酸、或者磷酸化的糖蛋白、抑或是C型凝集素Langerin发生结合, 之后在G蛋白偶联受体FFAR2、表达于膜上的核仁蛋白、适配体蛋白Epsin1和EPS15、C型凝集素Langerin等细胞因子的参与下, 通过网格蛋白介导的方式发生内吞. 胞内Ca2+的增加可以促进流感病毒通过网格蛋白或非网格蛋白的方式侵入细胞, 另外膜上脂筏结构也参与病毒的吸附和内化过程
Fig. 2. Schematic diagram of influenza virus binding and internalization at cellular surface. The influenza virus binds on the cell surface with sialic acid at the end of a glycoprotein or lipid, phosphorylated glycoprotein, or C-type lectin Langerin, and then enters into the cell through clathrin-mediated endocytosis with the participation of cytokines such as G protein coupled receptor FFAR2, nucleolus protein expressed in membrane, adapter proteins Epsin1 and EPS15, or C-type lectin Langerin. An increased amount of intracellular Ca2+ promotes the entry of influenza virus in the clathrin or non-clathrin way. Lipid rafts also play roles in the process of virus adsorption and internalization.
表 1 流感病毒与宿主细胞结合的靶向分子
Table 1. Targeting molecules for influenza virus binding to host cells.
表 2 参与流感病毒内化的细胞因子
Table 2. Cytokines involved in influenza virus internali-zation.
-
[1] Sieczkarski S B, Whittaker G R 2002 J. Gen. Virol. 83 1535
 Google Scholar
Google Scholar
[2] Nabi I R, Le P U 2003 J. Cell Biol. 161 673
 Google Scholar
Google Scholar
[3] Seto W H, Tsang D, Yung R W H, Ching T Y, Ng T K, Ho M, Ho L M, Peiris J S M, Auth A E S G H 2003 Lancet 361 1519
 Google Scholar
Google Scholar
[4] Wenzel R P, Edmond M B 2009 N. Engl. J. Med. 361 1991
 Google Scholar
Google Scholar
[5] de Groot R J, Baker S C, Baric R S, Brown C S, Drosten C, Enjuanes L, Fouchier R A M, Galiano M, Gorbalenya A E, Memish Z A, Perlman S, Poon L L M, Snijder E J, Stephens G M, Woo P C Y, Zaki A M, Zambon M, Ziebuhr J 2013 J. Virol. 87 7790
 Google Scholar
Google Scholar
[6] Gao R B, Cao B, Hu Y W, et al. 2013 N. Engl. J. Med. 368 1888
 Google Scholar
Google Scholar
[7] Song F X, Shi N N, Shan F, Zhang Z Y, Shen J, Lu H Z, Ling Y, Jiang Y B, Shi Y X 2020 Radiology 295 210
 Google Scholar
Google Scholar
[8] Li M, Ding H M, Lin M H, Yin F F, Song L, Mao X H, Li F, Ge Z L, Wang L H, Zuo X L, Ma Y Q, Fan C H 2019 J. Am. Chem. Soc. 141 18910
 Google Scholar
Google Scholar
[9] Wang H, Jiang C 2009 Sci. China. C Life Sci. 52 464
 Google Scholar
Google Scholar
[10] Lakadamyali M, Rust M J, Babcock H P, Zhuang X 2003 Proc. Natl. Acad. Sci. U. S. A. 100 9280
 Google Scholar
Google Scholar
[11] Fontana J, Steven A C 2013 J. Virol. 87 5621
 Google Scholar
Google Scholar
[12] Liu S L, Zhang Z L, Tian Z Q, Zhao H S, Liu H, Sun E Z, Xiao G F, Zhang W, Wang H Z, Pang D W 2011 ACS Nano 6 141
 Google Scholar
Google Scholar
[13] Babcock H P, Chen C, Zhuang X W 2004 Biophys. J. 87 2749
 Google Scholar
Google Scholar
[14] Wang M, Cao R, Zhang L, Yang X, Liu J, Xu M, Shi Z, Hu Z, Zhong W, Xiao G 2020 Cell Res. 30 269
 Google Scholar
Google Scholar
[15] Lu H 2020 Biosci. Trends 14 69
 Google Scholar
Google Scholar
[16] Li R F, Hou Y L, Huang J C, Pan W Q, Ma Q H, Shi Y X, Li C F, Zhao J, Jia Z H, Jiang H M, Zheng K, Huang S X, Dai J, Li X B, Hou X T, Wang L, Zhong N A, Yang Z F 2020 Pharmacol. Res. 156 104761
 Google Scholar
Google Scholar
[17] Dadonaite B, Vijayakrishnan S, Fodor E, Bhella D, Hutchinson E C 2016 J. Gen. Virol. 97 1755
 Google Scholar
Google Scholar
[18] Mosley V M, Wyckoff R W G 1946 Nature 157 263
 Google Scholar
Google Scholar
[19] Taylor A R, Sharp D G, Beard D, Beard J W, Dingle J H, Feller A E 1943 J. Immunol. 47 261
 Google Scholar
Google Scholar
[20] Sharp D G, Taylor A R, McLean I W, Jr Beard D, Beard J W, Fellew A E, Dingle J H 1943 Science 98 307
 Google Scholar
Google Scholar
[21] Choppin P W, Murphy J S, Tamm I 1960 J. Exp. Med. 112 945
 Google Scholar
Google Scholar
[22] Kilbourne E D, Murphy J S 1960 J. Exp. Med. 111 387
 Google Scholar
Google Scholar
[23] Kumlin U, Olofsson S, Dimock K, Arnberg N 2008 Influenza Other Resp. 2 147
 Google Scholar
Google Scholar
[24] Rogers G N, D'Souza B L 1989 Virology 173 317
 Google Scholar
Google Scholar
[25] Connor R J, Kawaoka Y, Webster R G, Paulson J C 1994 Virology 205 17
 Google Scholar
Google Scholar
[26] Peng W J, de Vries R P, Grant O C, Thompson A J, McBride R, Tsogtbaatar B, Lee P S, Razi N, Wilson I A, Woods R J, Paulson J C 2017 Cell Host Microbe 21 23
 Google Scholar
Google Scholar
[27] Ng W C, Liong S, Tate M D, Irimura T, Denda-Nagai K, Brooks A G, Londrigan S L, Reading P C 2014 J. Virol. 88 1659
 Google Scholar
Google Scholar
[28] Goronzy I N, Rawle R J, Boxer S G, Kasson P M 2018 Chem. Sci. 9 2340
 Google Scholar
Google Scholar
[29] Ding H M, Li J, Chen N, Hu X J, Yang X F, Guo L J, Li Q, Zuo X L, Wang L H, Ma Y Q, Fan C H 2018 Acs Central Sci 4 1344
 Google Scholar
Google Scholar
[30] Verma D K, Gupta D, Lal S K 2018 Viruses 10 650
 Google Scholar
Google Scholar
[31] Byrd-Leotis L, Jia N, Dutta S, Trost J F, Gao C, Cummings S F, Braulke T, Muller-Loennies S, Heimburg-Molinaro J, Steinhauer D A, Cummings R D 2019 Sci. Adv. 5 eaav2554
 Google Scholar
Google Scholar
[32] Ng W C, Londrigan S L, Nasr N, Cunningham A L, Turville S, Brooks A G, Reading P C 2016 J. Virol. 90 206
 Google Scholar
Google Scholar
[33] Oh N, Park J H 2014 Int. J. Nanomed. 9 51
 Google Scholar
Google Scholar
[34] Geiser M 2010 J. Aerosol Med. Pulm. Drug Deliv. 23 207
 Google Scholar
Google Scholar
[35] Dourmashkin R R, Tyrrell D A 1974 J. Gen. Virol. 24 129
 Google Scholar
Google Scholar
[36] Matlin K S, Reggio H, Helenius A, Simons K 1981 J. Cell Biol. 91 601
 Google Scholar
Google Scholar
[37] Conner S D, Schmid S L 2003 Nature 422 37
 Google Scholar
Google Scholar
[38] Nichols B J, Lippincott-Schwartz J 2001 Trends Cell Biol. 11 406
 Google Scholar
Google Scholar
[39] Li Y, Yue T T, Yang K, Zhang X R 2012 Biomaterials 33 4965
 Google Scholar
Google Scholar
[40] Rust M J, Lakadamyali M, Zhang F, Zhuang X W 2004 Nat. Struct. Mol. Biol. 11 567
 Google Scholar
Google Scholar
[41] Sieczkarski S B, Whittaker G R 2002 J. Virol. 76 10455
 Google Scholar
Google Scholar
[42] Sun E Z, Liu A A, Zhang Z L, Liu S L, Tian Z Q, Pang D W 2017 ACS Nano 11 4395
 Google Scholar
Google Scholar
[43] Thomsen P, Roepstorff K, Stahlhut M, van Deurs B, Riezman H 2002 Mol. Biol. Cell 13 238
 Google Scholar
Google Scholar
[44] Schnitzer J E, Oh P, Pinney E, Allard J 1994 J. Cell Biol. 127 1217
 Google Scholar
Google Scholar
[45] Orlandi P A, Fishman P H 1998 J. Cell Biol. 141 905
 Google Scholar
Google Scholar
[46] Nunes-Correia I, Eulalio A, Nir S, De Lima M C P 2004 Cell. Mol. Biol. Lett. 9 47
[47] Mercer J, Helenius A 2009 Nat. Cell Biol. 11 510
 Google Scholar
Google Scholar
[48] de Vries E, Tscherne D M, Wienholts M J, Cobos-Jimenez V, Scholte F, Garcia-Sastre A, Rottier P J, de Haan C A 2011 PLoS Pathog. 7 e1001329
 Google Scholar
Google Scholar
[49] Rossman J S, Leser G P, Lamb R A 2012 J. Virol. 86 10950
 Google Scholar
Google Scholar
[50] Carroll S M, Paulson J C 1985 Virus Res. 3 165
 Google Scholar
Google Scholar
[51] Williams S P, Robertson J S 1993 Virology 196 660
 Google Scholar
Google Scholar
[52] de Vries E, de Vries R P, Wienholts M J, Floris C E, Jacobs M S, van den Heuvel A, Rottier P J, de Haan C A 2012 Proc. Natl. Acad. Sci. U. S. A. 109 7457
 Google Scholar
Google Scholar
[53] Conner S D, Schmid S L 2003 J. Cell Biol. 162 773
 Google Scholar
Google Scholar
[54] Wang G, Jiang L, Wang J, Zhang J, Kong F, Li Q, Yan Y, Huang S, Zhao Y, Liang L, Li J, Sun N, Hu Y, Shi W, Deng G, Chen P, Liu L, Zeng X, Tian G, Bu Z, Chen H, Li C 2020 J. Virol. 94
 Google Scholar
Google Scholar
[55] Chen C, Zhuang X W 2008 Proc. Natl. Acad. Sci. U.S.A. 105 11790
 Google Scholar
Google Scholar
[56] Tayyari F, Marchant D, Moraes T J, Duan W M, Mastrangelo P, Hegele R G 2011 Nat. Med. 17 1132
 Google Scholar
Google Scholar
[57] Melen K, Tynell J, Fagerlund R, Roussel P, Hernandez-Verdun D, Julkunen I 2012 Virol J. 9 167
 Google Scholar
Google Scholar
[58] Murayama R, Harada Y, Shibata T, Kuroda K, Hayakawa S, Shimizu K, Tanaka T 2007 Biochem. Biophys. Res. Commun. 362 880
 Google Scholar
Google Scholar
[59] Chan C M, Chu H, Zhang A J, Leung L H, Sze K H, Kao R Y, Chik K K, To K K, Chan J F, Chen H, Jin D Y, Liu L, Yuen K Y 2016 Virology 494 78
 Google Scholar
Google Scholar
[60] Ji Q J, Yuan B, Lu X M, Yang K, Ma Y Q 2016 Small 12 1140
 Google Scholar
Google Scholar
[61] Geijtenbeek T B H, Gringhuis S I 2009 Nat. Rev. Immunol. 9 465
 Google Scholar
Google Scholar
[62] van Kooyk Y 2008 Biochem. Soc. Trans. 36 1478
 Google Scholar
Google Scholar
[63] Robinson M J, Sancho D, Slack E C, LeibundGut-Landmann S, Sousa C R E 2006 Nat. Immunol. 7 1258
 Google Scholar
Google Scholar
[64] Drickamer K 1992 Nature 360 183
 Google Scholar
Google Scholar
[65] Reading P C, Miller J L, Anders E M 2000 J. Virol. 74 5190
 Google Scholar
Google Scholar
[66] Valladeau J, Ravel O, Dezutter-Dambuyant C, Moore K, Kleijmeer M, Liu Y, Duvert-Frances V, Vincent C, Schmitt D, Davoust J, Caux C, Lebecque S, Saeland S 2000 Immunity 12 71
 Google Scholar
Google Scholar
[67] Cohen G B, Ren R B, Baltimore D 1995 Cell 80 237
 Google Scholar
Google Scholar
[68] Fujioka Y, Tsuda M, Nanbo A, Hattori T, Sasaki J, Sasaki T, Miyazaki T, Ohba Y 2013 Nat Commun 4 2763
 Google Scholar
Google Scholar
[69] Fujioka Y, Nishide S, Ose T, et al. 2018 Cell Host Microbe 23 809
 Google Scholar
Google Scholar
[70] Ehrhardt C, Marjuki H, Wolff T, Nuernberg B, Planz O, Pleschka S, Ludwig S 2006 Cell. Microbiol. 8 1336
 Google Scholar
Google Scholar
[71] Zhu L, Ly H, Liang Y 2014 J. Virol. 88 417
 Google Scholar
Google Scholar
[72] Chu V C, Whittaker G R 2004 Proc. Natl. Acad. Sci. U. S. A. 101 18153
 Google Scholar
Google Scholar
[73] Lu X, Xu P P, Ding H M, Yu Y S, Huo D, Ma Y Q 2019 Nat Commun 10 4520
 Google Scholar
Google Scholar
[74] Hamilton B S, Whittaker G R, Daniel S 2012 Viruses 4 1144
 Google Scholar
Google Scholar
[75] Fodor E 2013 Acta Virol. 57 113
 Google Scholar
Google Scholar
[76] Akarsu H, Burmeister W P, Petosa C, Petit I, Muller C W, Ruigrok R W H, Baudin F 2003 EMBO J. 22 4646
 Google Scholar
Google Scholar
[77] Dou D, Revol R, Ostbye H, Wang H, Daniels R 2018 Front Immunol 9 1581
 Google Scholar
Google Scholar
[78] Zhang J, Pekosz A, Lamb R A 2000 J. Virol. 74 4634
 Google Scholar
Google Scholar
[79] Dahmani I, Ludwig K, Chiantia S 2019 Biosci. Rep. 39 16
 Google Scholar
Google Scholar
[80] Pillay T S 2020 J. Clin. Pathol. 73 366
 Google Scholar
Google Scholar
[81] Han Q, Lin Q, Jin S, You L 2020 J. Infect. 80 373
 Google Scholar
Google Scholar
[82] Zhu N, Zhang D, Wang W, Li X, Yang B, Song J, Zhao X, Huang B, Shi W, Lu R, Niu P, Zhan F, Ma X, Wang D, Xu W, Wu G, Gao G F, Tan W, China Novel Coronavirus I, Research T 2020 N. Engl. J. Med. 382 727
 Google Scholar
Google Scholar
[83] Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, Wang W, Song H, Huang B, Zhu N, Bi Y, Ma X, Zhan F, Wang L, Hu T, Zhou H, Hu Z, Zhou W, Zhao L, Chen J, Meng Y, Wang J, Lin Y, Yuan J, Xie Z, Ma J, Liu W J, Wang D, Xu W, Holmes E C, Gao G F, Wu G, Chen W, Shi W, Tan W 2020 Lancet 395 565
 Google Scholar
Google Scholar
[84] Hoffmann M, Kleine-Weber H, Schroeder S, Kruger N, Herrler T, Erichsen S, Schiergens T S, Herrler G, Wu N H, Nitsche A, Muller M A, Drosten C, Pohlmann S 2020 Cell 181 271
 Google Scholar
Google Scholar
[85] Wang Q, Qiu Y, Li J Y, Zhou Z J, Liao C H, Ge X Y 2020 Virol. Sin. 35 337
 Google Scholar
Google Scholar
[86] Cheng Z, Zhou J, To K K, Chu H, Li C, Wang D, Yang D, Zheng S, Hao K, Bosse Y, Obeidat M, Brandsma C A, Song Y Q, Chen Y, Zheng B J, Li L, Yuen K Y 2015 J. Infect. Dis. 212 1214
 Google Scholar
Google Scholar
[87] Offringa A, Montijn R, Singh S, Paul M, Pinto Y M, Pinto-Sietsma S J 2020 Eur Heart J Cardiovasc Pharmacother pvaa053 doi: 10.1093/ehjcvp/pvaa053
[88] Sohail A, Nutini A 2020 Prog. Biophys. Mol. Biol. 155 29
 Google Scholar
Google Scholar
[89] Ng M L, Tan S H, See E E, Ooi E E, Ling A E 2003 J. Med. Virol. 71 323
 Google Scholar
Google Scholar
[90] Yang Z Y, Huang Y, Ganesh L, Leung K, Kong W P, Schwartz O, Subbarao K, Nabel G J 2004 J. Virol. 78 5642
 Google Scholar
Google Scholar
[91] Huang I C, Bosch B J, Li F, Li W H, Lee K H, Ghiran S, Vasilieva N, Dermody T S, Harrison S C, Dormitzer P R, Farzan M, Rottier P J M, Choe H 2006 J. Biol. Chem. 281 3198
 Google Scholar
Google Scholar
[92] Inoue Y, Tanaka N, Tanaka Y, Inoue S, Morita K, Zhuang M, Hattori T, Sugamura K 2007 J. Virol. 81 8722
 Google Scholar
Google Scholar
[93] Wang H L, Yang P, Liu K T, Guo F, Zhang Y L, Zhang G Y, Jiang C Y 2008 Cell Res. 18 290
 Google Scholar
Google Scholar
[94] Hofmann H, Pohlmann S 2004 Trends Microbiol. 12 466
 Google Scholar
Google Scholar
计量
- 文章访问数: 33210
- PDF下载量: 374
- 被引次数: 0




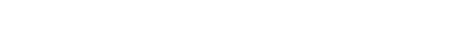








 下载:
下载: