-
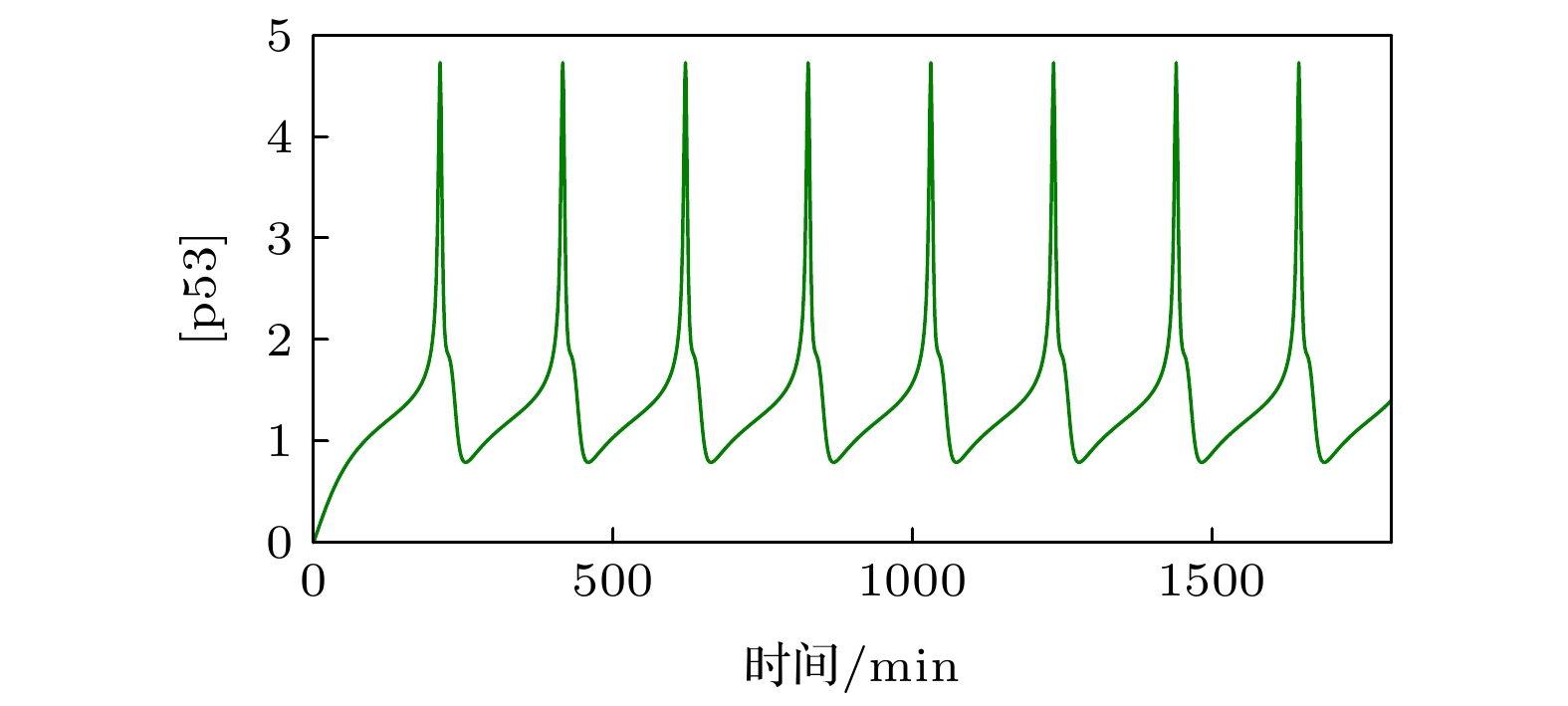
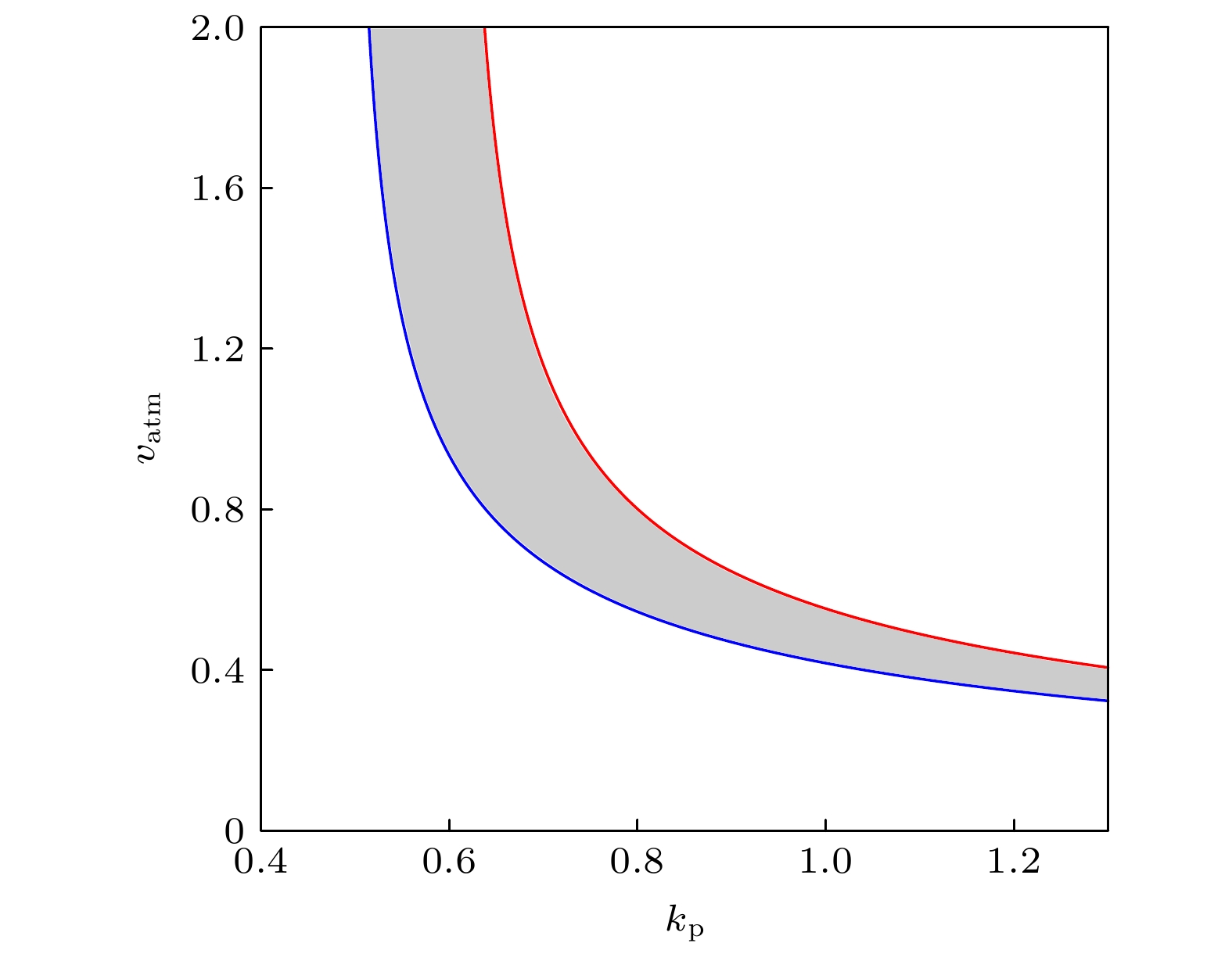
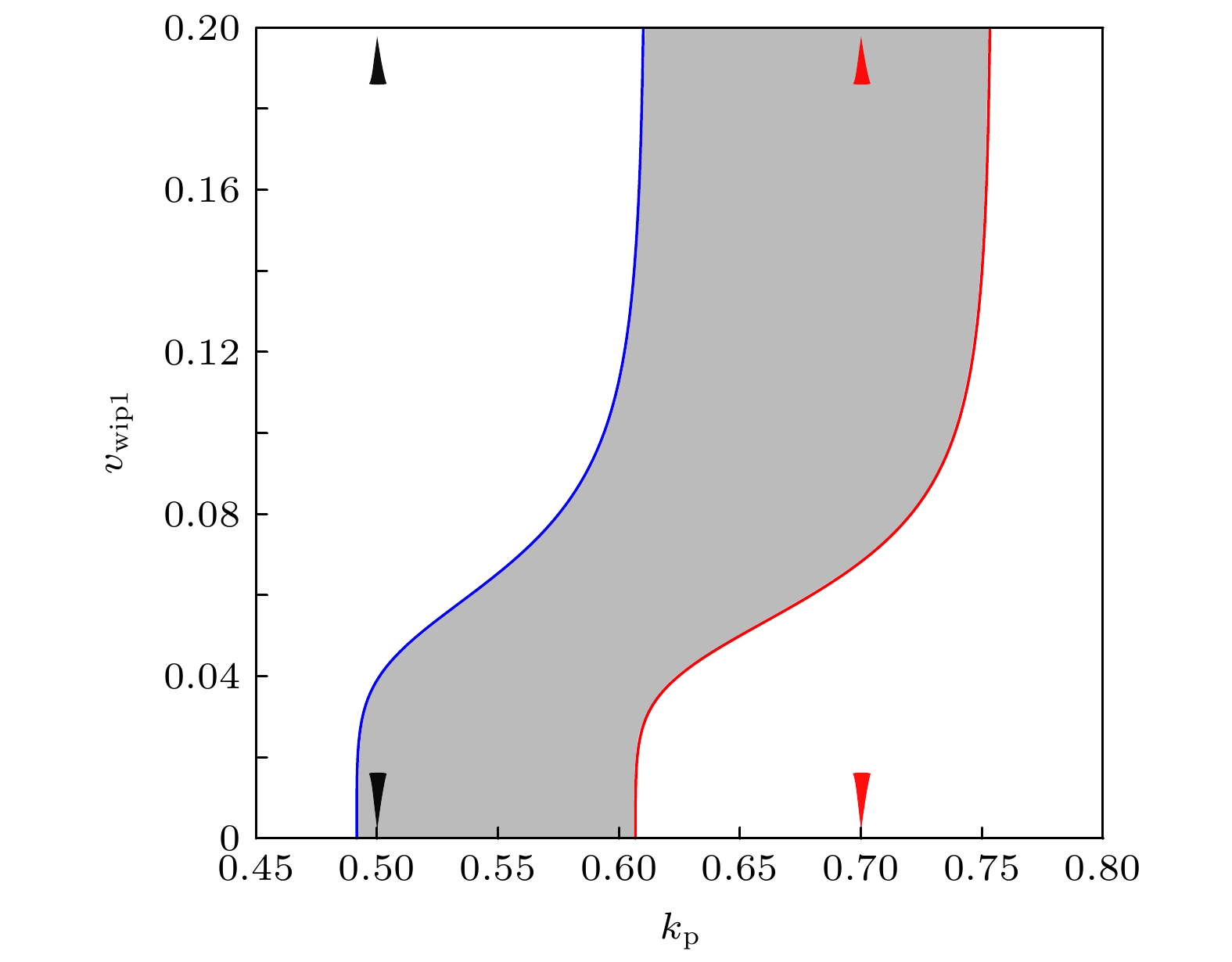
转录因子p53是细胞应激网络的核心, 以动态响应的方式控制基因毒性压力下的细胞命运抉择. Mdm2是种E3泛素连接酶, 既能破坏p53的稳定性又能提高p53的生成效率. Mdm2对p53的抑制性功能在p53-Mdm2振子中扮演着构建性角色, 而Mdm2对p53的促进性功能如何调控这个基因网络的动力学仍缺少研究. 因此, 本文利用数学模型, 全面探究了Mdm2上调p53的这条通路对p53动力学的影响. 结果表明: Mdm2在Ser395位点的磷酸化作用对p53的振荡必不可少; 之前报道的磷酸酶Wip1被p53振荡所需要, 可能仅发生在Mdm2所介导的正反馈通路强度较高的情景下; Mdm2促进p53的失活以及泛素化降解也是p53反复振动动力学发生的关键因素, 与以往的结论一致. 本文的结果可对今后p53动力学的相关实验起到一定的指导作用.The transcription factor p53 is the core of the cellular stress network, which controls cell fate decisions on genotoxic stress in a dynamics response manner. Mdm2 is an E3 ubiquitin ligase, which play a dual role in p53 regulation, that is, on the one hand, Mdm2 destroys the stability of p53 via ubiquitin labeling, and on the other hand, Mdm2 increase the efficiency of p53 production through binding p53 messenger RNA. A number of models indicate that Mdm2's inhibitory function on p53 plays a constructive role in the p53-Mdm2 vibrator, the research of how Mdm2's promotion function on p53 regulates the dynamics of this gene network, however, is still lacking. To this end, this paper uses a mathematical model to comprehensively explore the influence of the Mdm2 up-regulation p53 pathway on p53 dynamics. We adopt the ordinary differential equations (ODE) model and the bifurcation analysis method to investigate the multiple role of Mdm2 on p53 gene networks dynamics control. The results show that the phosphorylation of Mdm2 at Ser395, which forming a positive feedback loop (PFL) with p53, is essential for the oscillation of this gene network and may be the reason why there is a bimodal switch of p53 kinetics under etoposide stimulation; the previously reported phosphatase Wip1 is required for the oscillation of p53, which may only occur in situations where the intensity of the PFL mediated by phosphorylated Mdm2 is high, and we predict that Wip1 will inhibit the p53 oscillation in some weak PFL cases; Finally, our model also proves that Mdm2 promotes the deactivation and ubiquitination degradation of p53 is a key factor in the occurrence of repeated vibration dynamics of p53. The main conclusions of this paper are exhibited in some two-dimensional bifurcation diagrams. In order to confirm these bifurcation phenomena, we discuss the case where the Hill coefficient describing the degree of nonlinearity is small. As a result, there is no obvious shape change of the bifurcation curves surrounding the oscillation region. The robustness of these conclusions is verified again. We believe that our results can play a guiding role in the future related experiments of p53 kinetics.
-
Keywords:
- Mdm2 /
- p53 /
- feedback /
- bifurcation
[1] 李翔, 刘锋, 帅建伟 2016 物理学报 65 178704
 Google Scholar
Google Scholar
Li X, Liu F, Shuai J W 2016 Acta Phys. Sin. 65 178704
 Google Scholar
Google Scholar
[2] Puszyński K, Hat B, Lipniacki T 2008 J. Theor. Biol. 254 452
 Google Scholar
Google Scholar
[3] Kubbutat M H G, Jones S N, Vousden K H 1997 Nature 387 299
 Google Scholar
Google Scholar
[4] Sermeus A, Michiels C 2011 Cell Death Dis. 2 e164
 Google Scholar
Google Scholar
[5] Li H, Zhang X P, Liu F 2013 PLoS One 8 e80111
 Google Scholar
Google Scholar
[6] Wang P, Han B, Zhang X P, Liu F, Wang W 2018 FEBS Lett. 593 13
 Google Scholar
Google Scholar
[7] Shikazono N, Noguchi M, Fujii K, Urushibara A, Yokoya A 2009 J. Radiated Res. 50 27
 Google Scholar
Google Scholar
[8] Vousden K H 2006 J. Cell Sci. 119 5015
 Google Scholar
Google Scholar
[9] Purvis J E, Karhohs K W, Mock C, Batchelor E, Loewer A, Lahav G 2012 Science 336 1440
 Google Scholar
Google Scholar
[10] Lev Bar-Or R, Maya R, Segel L A, Alon U, Levine A J, Oren M 2000 Proc. Natl. Acad. Sci. U.S.A. 97 11250
 Google Scholar
Google Scholar
[11] Geva-Zatorsky N, Rosenfeld N, Itzkovitz S, Milo R, Sigal A, Dekel E, Yarnitzky T, Liron Y, Polak P, Lahav G, Alon U 2006 Mol. Syst. Biol. 2 2006.0033
 Google Scholar
Google Scholar
[12] Lahav G, Rosenfeld N, Sigal A, Geva-Zatorsky N, Levine A J, Elowitz M B, Alon U 2004 Nat. Genet. 36 147
 Google Scholar
Google Scholar
[13] 张丽娟, 晏世伟, 卓益忠 2007 物理学报 56 2442
 Google Scholar
Google Scholar
Zhang L J, Yan S W, Zhuo Y Z 2007 Acta Phys. Sin. 56 2442
 Google Scholar
Google Scholar
[14] Bottani S, Grammaticos B 2007 J. Theor. Biol. 249 235
 Google Scholar
Google Scholar
[15] Ma L, Wagner J, Rice J J, Hu W, Levine A J, Stolovitzky G 2005 Proc. Natl. Acad. Sci. U.S.A. 102 14266
 Google Scholar
Google Scholar
[16] Xia J F, Jia Y 2010 Chin. Phys. B 19 040506
 Google Scholar
Google Scholar
[17] Eliaš J, Dimitrio L, Clairambault J, Natalini P 2014 Phys. Biol. 11 045001
 Google Scholar
Google Scholar
[18] 毕远宏, 杨卓琴, 何小燕 2016 物理学报 65 028701
 Google Scholar
Google Scholar
Bi Y H, Yang Z Q, He X Y 2016 Acta Phys. Sin. 65 028701
 Google Scholar
Google Scholar
[19] Hat B, Kochańczyk M, Bogdal M N, Lipniacki T 2016 PLoS Comput. Biol. 12 e1004787
 Google Scholar
Google Scholar
[20] Wang D G, Zhou C H, Zhang X P 2017 Chin. Phys. B 26 128709
 Google Scholar
Google Scholar
[21] Ochab M, Puszynski K 2020 PLoS One 15 e0243823
 Google Scholar
Google Scholar
[22] Liu N, Wang D N, Liu H Y, Yang H L, Yang L G 2020 Chin. Phys. B 29 068704
 Google Scholar
Google Scholar
[23] Batchelor E, Loewer A, Mock C, Lahav J 2011 Mol. Syst. Biol. 7 488
 Google Scholar
Google Scholar
[24] Chen X, Chen J, Gan S, Guan H, Zhou Y, Ouyang Q, Shi J 2013 BMC Biol. 11 73
 Google Scholar
Google Scholar
[25] Falck J, Coates J, Jackson S P 2005 Nature 434 605
 Google Scholar
Google Scholar
[26] Stommel J, Wahl G 2004 EMBO J. 23 1547
 Google Scholar
Google Scholar
[27] Hamard P J, Manfredi J J 2012 Cancer Cell 21 3
 Google Scholar
Google Scholar
[28] Mayo L D, Donner D B 2001 Proc. Natl. Acad. Sci. U.S.A. 98 11598
 Google Scholar
Google Scholar
[29] Batchelor E, Mock C S, Bhan I, Loewer A, Lahav G 2008 Mol. Cell 30 277
 Google Scholar
Google Scholar
[30] Zhang X P, Liu F, Wang W 2011 Proc. Natl. Acad. Sci. U.S.A. 108 8990
 Google Scholar
Google Scholar
[31] Zhuge C, Sun X, Chen Y, Lei J 2016 J. Theor. Biol. 388 1
 Google Scholar
Google Scholar
[32] Sun T, Cui J 2014 FEBS Lett. 588 815
 Google Scholar
Google Scholar
[33] Zhang T, Brazhnik P, Tyson J J 2007 Cell Cycle 6 85
 Google Scholar
Google Scholar
[34] 王耀来, 刘锋 2020 物理学报 69 248702
 Google Scholar
Google Scholar
Wang Y L, Liu F 2020 Acta Phys. Sin. 69 248702
 Google Scholar
Google Scholar
[35] 曹志, 王卫东 2015 生物化学与生物物理进展 42 147
 Google Scholar
Google Scholar
Cao Z, Wang W D 2015 Prog. Biochem. Biophys. 42 147
 Google Scholar
Google Scholar
[36] Jeffrey P D, Gorina S, Pavletich N P 1995 Science 267 1498
 Google Scholar
Google Scholar
-
表 1 模型中的参数
Table 1. Parameters of the model
参数 参数描述 值 单位 $v_{\rm {atm}}$ ATM激活速率 1 ${\rm{C} }\!\cdot\! {\rm{min} }^{-1}$ $d_{\rm {atm}}$ ATM最大失活速率 0.53 ${\rm{min}}^{-1}$ $\rho_ 0$ Wip1依赖的ATM失活反应占据的比例常数 0.9 $\setminus$ $k_ 0$ Wip1催化的ATM失活反应中的酶半饱和浓度 1 ${\rm{C}}$ $v_ {\rm{p53}}$ p53最大合成并激活的速率 0.95 ${\rm{C} }\!\cdot\! {\rm{min} }^{-1}$ $\rho_ 1$ ATM依赖的p53激活反应占据的比例常数 0.9 $\setminus$ $k_ 1$ ATM催化的p53激活反应中的酶半饱和浓度 0.3 ${\rm{C}}$ $\rho_ 2$ Mdm2p1依赖的p53合成反应占据的比例常数 0.98 $\setminus$ $k_ 2$ Mdm2p1催化的p53合成反应中的酶半饱和浓度 0.057 ${\rm{C}}$ $d_ {\rm{p53}}$ p53最大降解速率 0.6 ${\rm{min}}^{-1}$ $\rho_ 3$ Mdm2p2依赖的p53失活和降解反应占据的比例常数 0.97 $\setminus$ $k_ 3$ Mdm2p2催化的p53失活和降解反应中的酶半饱和浓度 0.09 ${\rm{C}}$ $v_ {\rm{mdm2}}$ Mdm2最大生成速率 0.153 ${\rm{C} }\!\cdot\! {\rm{min} }^{-1}$ $\rho_ 4$ p53依赖的Mdm2合成反应占据的比例常数 0.98 $\setminus$ $k_ 4$ p53催化的Mdm2合成反应中的酶半饱和浓度 4.43 ${\rm{C}}$ $k_ {\rm{p}}$ Mdm2在Ser395位点的最大磷酸化速率 0.65 ${\rm{min}}^{-1}$ $\rho_ 5$ ATM依赖的Mdm2磷酸化反应占据的比例常数 0.9 $\setminus$ $k_ 5$ ATM催化的Mdm2磷酸化反应中的酶半饱和浓度 1 ${\rm{C}}$ $k_{\rm{ q}}$ Mdm2在Ser395位点的去磷酸化速率 0.24 ${\rm{min}}^{-1}$ ${k_ {{\rm{p}}*}}$ Mdm2在Ser166, Ser168的磷酸化速率 0.14 ${\rm{min}}^{-1}$ ${k_ {{\rm{q}}*}}$ Mdm2在Ser166, Ser168的去磷酸化速率 0.01 ${\rm{min}}^{-1}$ $d_ {\rm{mdm2}}$ Mdm2本底降解速率 0.034 ${\rm{min}}^{-1}$ $v_ {\rm{wip1}}$ Wip1最大生成速率 0.09 ${\rm{C} }\!\cdot\! {\rm{min} }^{-1}$ $\rho_ 6$ p53依赖的Wip1合成反应占据的比例常数 0.99 $\setminus$ $k_ 6$ p53催化的Wip1合成反应中的酶半饱和浓度 0.8 ${\rm{C}}$ $d_ {\rm{wip1}}$ Wip1降解速率 0.05 ${\rm{min}}^{-1}$ f Mdm2p1的降解速率与Mdm2本底降解速率的比值 3.58 $\setminus$ g Mdm2p2的降解速率与Mdm2本底降解速率的比值 1.95 $\setminus$ -
[1] 李翔, 刘锋, 帅建伟 2016 物理学报 65 178704
 Google Scholar
Google Scholar
Li X, Liu F, Shuai J W 2016 Acta Phys. Sin. 65 178704
 Google Scholar
Google Scholar
[2] Puszyński K, Hat B, Lipniacki T 2008 J. Theor. Biol. 254 452
 Google Scholar
Google Scholar
[3] Kubbutat M H G, Jones S N, Vousden K H 1997 Nature 387 299
 Google Scholar
Google Scholar
[4] Sermeus A, Michiels C 2011 Cell Death Dis. 2 e164
 Google Scholar
Google Scholar
[5] Li H, Zhang X P, Liu F 2013 PLoS One 8 e80111
 Google Scholar
Google Scholar
[6] Wang P, Han B, Zhang X P, Liu F, Wang W 2018 FEBS Lett. 593 13
 Google Scholar
Google Scholar
[7] Shikazono N, Noguchi M, Fujii K, Urushibara A, Yokoya A 2009 J. Radiated Res. 50 27
 Google Scholar
Google Scholar
[8] Vousden K H 2006 J. Cell Sci. 119 5015
 Google Scholar
Google Scholar
[9] Purvis J E, Karhohs K W, Mock C, Batchelor E, Loewer A, Lahav G 2012 Science 336 1440
 Google Scholar
Google Scholar
[10] Lev Bar-Or R, Maya R, Segel L A, Alon U, Levine A J, Oren M 2000 Proc. Natl. Acad. Sci. U.S.A. 97 11250
 Google Scholar
Google Scholar
[11] Geva-Zatorsky N, Rosenfeld N, Itzkovitz S, Milo R, Sigal A, Dekel E, Yarnitzky T, Liron Y, Polak P, Lahav G, Alon U 2006 Mol. Syst. Biol. 2 2006.0033
 Google Scholar
Google Scholar
[12] Lahav G, Rosenfeld N, Sigal A, Geva-Zatorsky N, Levine A J, Elowitz M B, Alon U 2004 Nat. Genet. 36 147
 Google Scholar
Google Scholar
[13] 张丽娟, 晏世伟, 卓益忠 2007 物理学报 56 2442
 Google Scholar
Google Scholar
Zhang L J, Yan S W, Zhuo Y Z 2007 Acta Phys. Sin. 56 2442
 Google Scholar
Google Scholar
[14] Bottani S, Grammaticos B 2007 J. Theor. Biol. 249 235
 Google Scholar
Google Scholar
[15] Ma L, Wagner J, Rice J J, Hu W, Levine A J, Stolovitzky G 2005 Proc. Natl. Acad. Sci. U.S.A. 102 14266
 Google Scholar
Google Scholar
[16] Xia J F, Jia Y 2010 Chin. Phys. B 19 040506
 Google Scholar
Google Scholar
[17] Eliaš J, Dimitrio L, Clairambault J, Natalini P 2014 Phys. Biol. 11 045001
 Google Scholar
Google Scholar
[18] 毕远宏, 杨卓琴, 何小燕 2016 物理学报 65 028701
 Google Scholar
Google Scholar
Bi Y H, Yang Z Q, He X Y 2016 Acta Phys. Sin. 65 028701
 Google Scholar
Google Scholar
[19] Hat B, Kochańczyk M, Bogdal M N, Lipniacki T 2016 PLoS Comput. Biol. 12 e1004787
 Google Scholar
Google Scholar
[20] Wang D G, Zhou C H, Zhang X P 2017 Chin. Phys. B 26 128709
 Google Scholar
Google Scholar
[21] Ochab M, Puszynski K 2020 PLoS One 15 e0243823
 Google Scholar
Google Scholar
[22] Liu N, Wang D N, Liu H Y, Yang H L, Yang L G 2020 Chin. Phys. B 29 068704
 Google Scholar
Google Scholar
[23] Batchelor E, Loewer A, Mock C, Lahav J 2011 Mol. Syst. Biol. 7 488
 Google Scholar
Google Scholar
[24] Chen X, Chen J, Gan S, Guan H, Zhou Y, Ouyang Q, Shi J 2013 BMC Biol. 11 73
 Google Scholar
Google Scholar
[25] Falck J, Coates J, Jackson S P 2005 Nature 434 605
 Google Scholar
Google Scholar
[26] Stommel J, Wahl G 2004 EMBO J. 23 1547
 Google Scholar
Google Scholar
[27] Hamard P J, Manfredi J J 2012 Cancer Cell 21 3
 Google Scholar
Google Scholar
[28] Mayo L D, Donner D B 2001 Proc. Natl. Acad. Sci. U.S.A. 98 11598
 Google Scholar
Google Scholar
[29] Batchelor E, Mock C S, Bhan I, Loewer A, Lahav G 2008 Mol. Cell 30 277
 Google Scholar
Google Scholar
[30] Zhang X P, Liu F, Wang W 2011 Proc. Natl. Acad. Sci. U.S.A. 108 8990
 Google Scholar
Google Scholar
[31] Zhuge C, Sun X, Chen Y, Lei J 2016 J. Theor. Biol. 388 1
 Google Scholar
Google Scholar
[32] Sun T, Cui J 2014 FEBS Lett. 588 815
 Google Scholar
Google Scholar
[33] Zhang T, Brazhnik P, Tyson J J 2007 Cell Cycle 6 85
 Google Scholar
Google Scholar
[34] 王耀来, 刘锋 2020 物理学报 69 248702
 Google Scholar
Google Scholar
Wang Y L, Liu F 2020 Acta Phys. Sin. 69 248702
 Google Scholar
Google Scholar
[35] 曹志, 王卫东 2015 生物化学与生物物理进展 42 147
 Google Scholar
Google Scholar
Cao Z, Wang W D 2015 Prog. Biochem. Biophys. 42 147
 Google Scholar
Google Scholar
[36] Jeffrey P D, Gorina S, Pavletich N P 1995 Science 267 1498
 Google Scholar
Google Scholar
计量
- 文章访问数: 7493
- PDF下载量: 82
- 被引次数: 0













 下载:
下载: