-
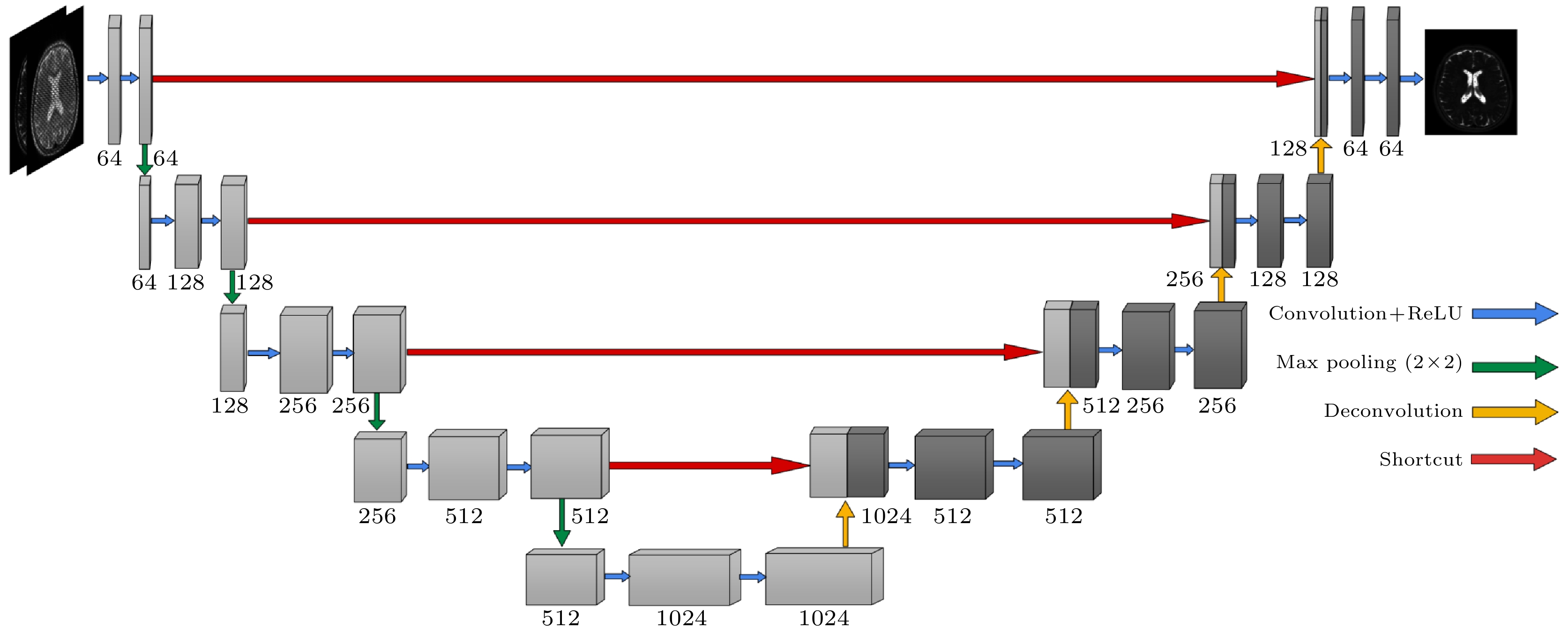
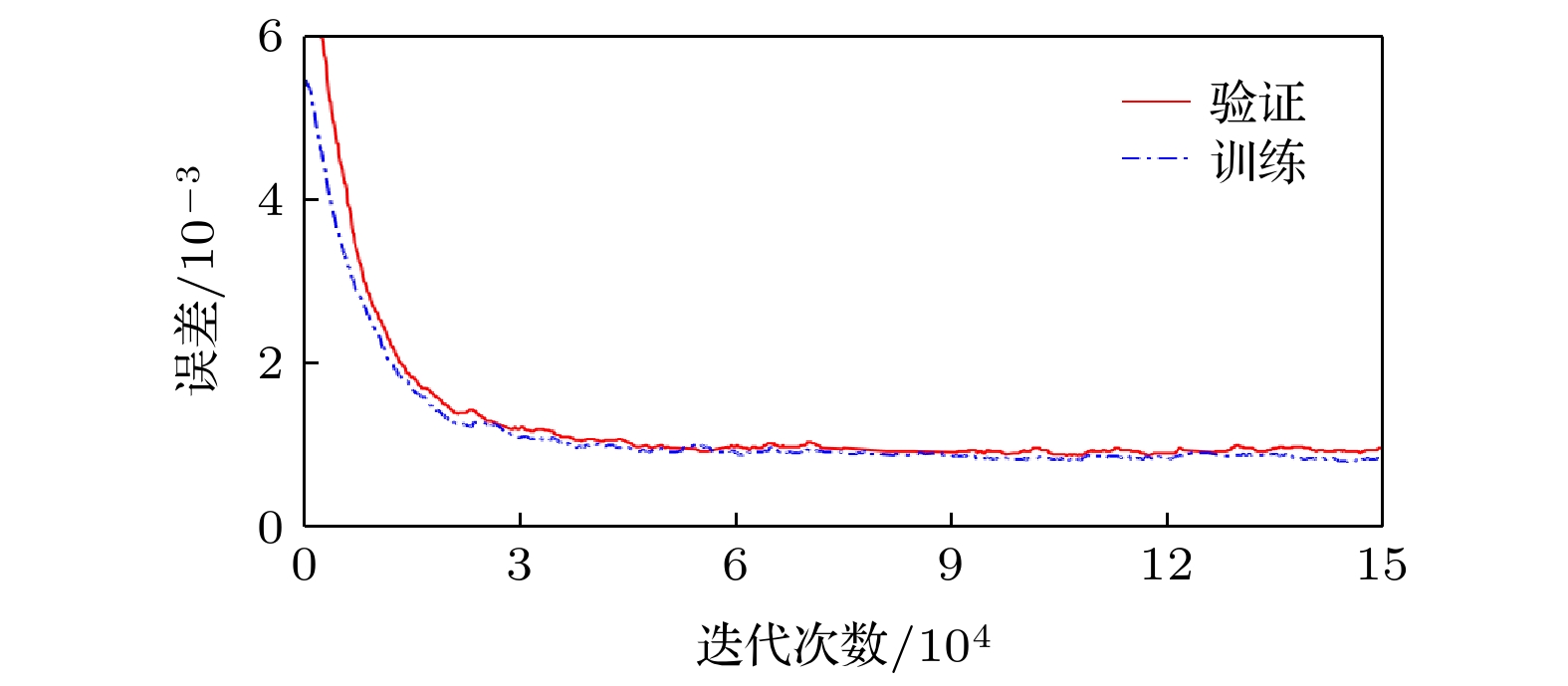
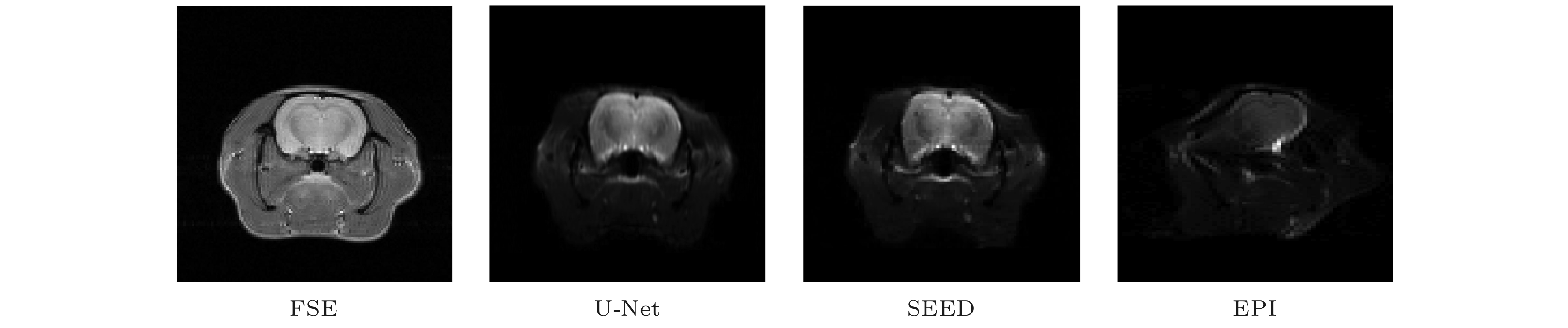
单扫描时空编码磁共振成像是一种新型超快速磁共振成像技术, 它对磁场不均匀和化学位移伪影有较强的抵抗性, 但是其固有的空间分辨率较低, 因此通常需要进行超分辨率重建, 以在不增加采样点数的情况下提高时空编码磁共振图像的空间分辨率. 然而, 现有的重建方法存在迭代求解时间长、重建结果有混叠伪影残留等问题. 为此, 本文提出了一种基于深度神经网络的单扫描时空编码磁共振成像超分辨率重建方法. 该方法采用模拟样本训练深度神经网络, 再利用训练好的网络模型对实际采样信号进行重建. 数值模拟、水模和活体鼠脑的实验结果表明, 该方法能快速重建出无残留混叠伪影、纹理信息清楚的超分辨率时空编码磁共振图像. 适当增加训练样本数量以及在训练样本中加入适当的随机噪声水平, 有助于改善重建效果.Single-shot spatiotemporally-encoded magnetic resonance imaging (SPEN MRI) is a novel ultrafast MRI technology. The SPEN MRI possesses great resistance to inhomogeneous B0 magnetic field and chemical shift effect. However, it has inherently low spatial resolution, and the super-resolved reconstruction is required to improve the spatial resolution of SPEN MRI image without additional signal acquisition. Several super-resolved reconstruction methods have been proposed, but they all suffer the problems of long iterative solution time and/or aliasing artifacts residue in the reconstructed results. In this paper, a super-resolved reconstruction method is proposed for single-shot SPEN MRI based on deep neural network. In this method the simulation samples are used to train the deep neural network, and then the trained network model is adopted to reconstruct the real sampled signals. Experimental results of numerical simulation, water phantom and in vivo rat brain show that this method can quickly reconstruct a super-resolved SPEN image with no residual aliasing artifacts, and clear texture information. An appropriate number of training samples and an appropriate random noise level for training samples contribute to improving the reconstruction results.
-
Keywords:
- magnetic resonance imaging /
- spatiotemporal encoding /
- super-resolved reconstruction /
- deep neural network
[1] Shrot Y, Frydman L 2005 J. Magn. Reson. 172 179
 Google Scholar
Google Scholar
[2] Tal A, Frydman L 2006 J. Magn. Reson. 182 179
 Google Scholar
Google Scholar
[3] Solomon E, Avni R, Hadas R, Raz T, Garbow J R, Bendel P, Frydman L, Neeman M 2014 Proc. Nati. Acad. Sci. USA 111 10353
 Google Scholar
Google Scholar
[4] Ben-Eliezer N, Irani M, Frydman L 2010 Magn. Reson. Med. 63 1594
 Google Scholar
Google Scholar
[5] Chen Y, Li J, Qu X B, Chen L, Cai C B, Cai S H, Zhong J H, Chen Z 2013 Magn. Reson. Med. 69 1326
 Google Scholar
Google Scholar
[6] Cai C B, Dong J Y, Cai S H, Li J, Chen Y, Bao L J, Chen Z 2013 J. Magn. Reson. 228 136
 Google Scholar
Google Scholar
[7] Chen L, Li J, Zhang M, Cai S H, Zhang T, Cai C B, Chen Z 2015 Med. Image Anal. 23 1
 Google Scholar
Google Scholar
[8] Aliotta E, Nourzadeh H, Sanders J, Muller D, Ennis D B 2019 Med. Phys. 46 1581
 Google Scholar
Google Scholar
[9] Chun J, Zhang H, Gach H M, Olberg S, Mazur T, Green O, Kim T, Kim H, Kim J S, Mutic S, Park J C 2019 Med. Phys. 46 4148
 Google Scholar
Google Scholar
[10] Le M H, Chen J, Wang L, Wang Z, Liu W, Cheng K T, Yang X 2017 Phys. Med. Biol. 62 6497
 Google Scholar
Google Scholar
[11] Liu Y, Lei Y, Wang Y, Wang T, Ren L, Lin L, McDonald M, Curran W J, Liu T, Zhou J, Yang X 2019 Phys. Med. Biol. 64 145015
 Google Scholar
Google Scholar
[12] 罗伶俐, 王远军 2020 中国医学物理学杂志 37 873
 Google Scholar
Google Scholar
Luo L L, Wang Y J 2020 Chin. J. Med. Phys. 37 873
 Google Scholar
Google Scholar
[13] 王甜甜, 王慧, 朱艳春, 王丽嘉 2021 物理学报 70 228701
 Google Scholar
Google Scholar
Wang T T, Wang H, Zhu Y C, Wang L J 2021 Acta Phys. Sin. 70 228701
 Google Scholar
Google Scholar
[14] Schlemper J, Caballero J, Hajnal J V, Price A N, Rueckert D 2018 IEEE Trans. Med. Imaging 37 491
 Google Scholar
Google Scholar
[15] Shi J, Liu Q, Wang C, Zhang Q, Ying S, Xu H 2018 Phys. Med. Biol. 63 085011
 Google Scholar
Google Scholar
[16] Quan T M, Nguyen-Duc T, Jeong W K 2018 IEEE Trans. Med. Imaging 37 1488
 Google Scholar
Google Scholar
[17] Guo C L, Wu J, Rosenberg J T, Roussel T, Cai S H, Cai C B 2020 Magn. Reson. Med. 84 3192
 Google Scholar
Google Scholar
[18] Akkus Z, Galimzianova A, Hoogi A, Rubin D L, Erickson B J 2017 J. Digit. Imaging 30 449
 Google Scholar
Google Scholar
[19] Zhang J, Wu J, Chen S J, Zhang Z Y, Cai S H, Cai C B, Chen Z 2019 IEEE Trans. Med. Imaging 38 1801
 Google Scholar
Google Scholar
[20] Ronneberger O, Fischer P, Brox T 2015 18th International Conference on Medical Image Computing and Computer-Assisted Intervention (MICCAI) Munich, Germany, October 5–9, 2015 p234
[21] Siddique N, Paheding S, Elkin C P, Devabhaktuni V 2021 IEEE Access 9 82031
 Google Scholar
Google Scholar
[22] Yang G, Yu S M, Dong H, Slabaugh G, Dragotti P L, Ye X J, Liu F D, Arridge S, Keegan J, Guo Y K, Firmin D 2018 IEEE Trans. Med. Imaging 37 1310
 Google Scholar
Google Scholar
[23] Liu F, Velikina J V, Block W F, Kijowski R, Samsonov A A 2017 IEEE Trans. Med. Imaging 36 527
 Google Scholar
Google Scholar
-
-
[1] Shrot Y, Frydman L 2005 J. Magn. Reson. 172 179
 Google Scholar
Google Scholar
[2] Tal A, Frydman L 2006 J. Magn. Reson. 182 179
 Google Scholar
Google Scholar
[3] Solomon E, Avni R, Hadas R, Raz T, Garbow J R, Bendel P, Frydman L, Neeman M 2014 Proc. Nati. Acad. Sci. USA 111 10353
 Google Scholar
Google Scholar
[4] Ben-Eliezer N, Irani M, Frydman L 2010 Magn. Reson. Med. 63 1594
 Google Scholar
Google Scholar
[5] Chen Y, Li J, Qu X B, Chen L, Cai C B, Cai S H, Zhong J H, Chen Z 2013 Magn. Reson. Med. 69 1326
 Google Scholar
Google Scholar
[6] Cai C B, Dong J Y, Cai S H, Li J, Chen Y, Bao L J, Chen Z 2013 J. Magn. Reson. 228 136
 Google Scholar
Google Scholar
[7] Chen L, Li J, Zhang M, Cai S H, Zhang T, Cai C B, Chen Z 2015 Med. Image Anal. 23 1
 Google Scholar
Google Scholar
[8] Aliotta E, Nourzadeh H, Sanders J, Muller D, Ennis D B 2019 Med. Phys. 46 1581
 Google Scholar
Google Scholar
[9] Chun J, Zhang H, Gach H M, Olberg S, Mazur T, Green O, Kim T, Kim H, Kim J S, Mutic S, Park J C 2019 Med. Phys. 46 4148
 Google Scholar
Google Scholar
[10] Le M H, Chen J, Wang L, Wang Z, Liu W, Cheng K T, Yang X 2017 Phys. Med. Biol. 62 6497
 Google Scholar
Google Scholar
[11] Liu Y, Lei Y, Wang Y, Wang T, Ren L, Lin L, McDonald M, Curran W J, Liu T, Zhou J, Yang X 2019 Phys. Med. Biol. 64 145015
 Google Scholar
Google Scholar
[12] 罗伶俐, 王远军 2020 中国医学物理学杂志 37 873
 Google Scholar
Google Scholar
Luo L L, Wang Y J 2020 Chin. J. Med. Phys. 37 873
 Google Scholar
Google Scholar
[13] 王甜甜, 王慧, 朱艳春, 王丽嘉 2021 物理学报 70 228701
 Google Scholar
Google Scholar
Wang T T, Wang H, Zhu Y C, Wang L J 2021 Acta Phys. Sin. 70 228701
 Google Scholar
Google Scholar
[14] Schlemper J, Caballero J, Hajnal J V, Price A N, Rueckert D 2018 IEEE Trans. Med. Imaging 37 491
 Google Scholar
Google Scholar
[15] Shi J, Liu Q, Wang C, Zhang Q, Ying S, Xu H 2018 Phys. Med. Biol. 63 085011
 Google Scholar
Google Scholar
[16] Quan T M, Nguyen-Duc T, Jeong W K 2018 IEEE Trans. Med. Imaging 37 1488
 Google Scholar
Google Scholar
[17] Guo C L, Wu J, Rosenberg J T, Roussel T, Cai S H, Cai C B 2020 Magn. Reson. Med. 84 3192
 Google Scholar
Google Scholar
[18] Akkus Z, Galimzianova A, Hoogi A, Rubin D L, Erickson B J 2017 J. Digit. Imaging 30 449
 Google Scholar
Google Scholar
[19] Zhang J, Wu J, Chen S J, Zhang Z Y, Cai S H, Cai C B, Chen Z 2019 IEEE Trans. Med. Imaging 38 1801
 Google Scholar
Google Scholar
[20] Ronneberger O, Fischer P, Brox T 2015 18th International Conference on Medical Image Computing and Computer-Assisted Intervention (MICCAI) Munich, Germany, October 5–9, 2015 p234
[21] Siddique N, Paheding S, Elkin C P, Devabhaktuni V 2021 IEEE Access 9 82031
 Google Scholar
Google Scholar
[22] Yang G, Yu S M, Dong H, Slabaugh G, Dragotti P L, Ye X J, Liu F D, Arridge S, Keegan J, Guo Y K, Firmin D 2018 IEEE Trans. Med. Imaging 37 1310
 Google Scholar
Google Scholar
[23] Liu F, Velikina J V, Block W F, Kijowski R, Samsonov A A 2017 IEEE Trans. Med. Imaging 36 527
 Google Scholar
Google Scholar
计量
- 文章访问数: 9492
- PDF下载量: 177
- 被引次数: 0













 下载:
下载: