-
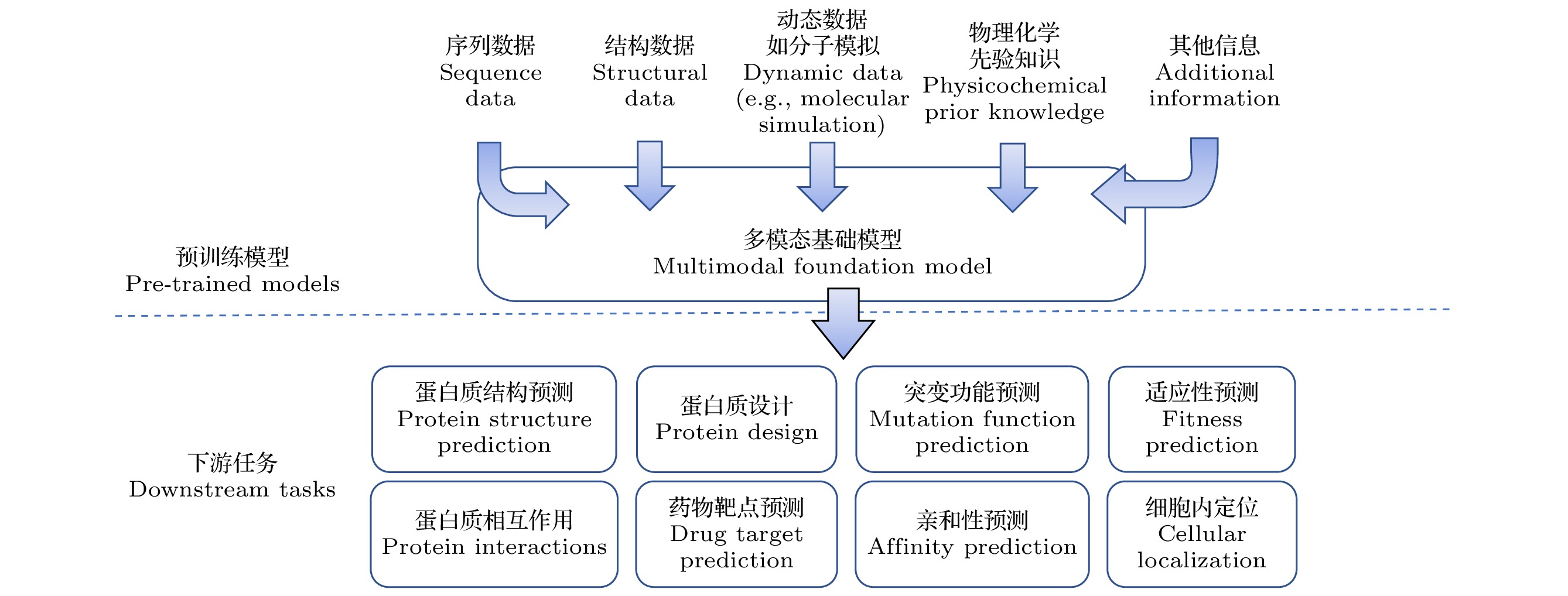
The AI revolution, sparked by natural language and image processing, has brought new ideas and research paradigms to the field of protein computing. One significant advancement is the development of pre-training protein language models through self-supervised learning from massive protein sequences. These pre-trained models encode various information about protein sequences, evolution, structures, and even functions, which can be easily transferred to various downstream tasks and demonstrate robust generalization capabilities. Recently, researchers have further developed multimodal pre-trained models that integrate more diverse types of data. The recent studies in this direction are summarized and reviewed from the following aspects in this paper. Firstly, the protein pre-training models that integrate protein structures into language models are reviewed: this is particularly important, for protein structure is the primary determinant of its function. Secondly, the pre-trained models that integrate protein dynamic information are introduced. These models may benefit downstream tasks such as protein-protein interactions, soft docking of ligands, and interactions involving allosteric proteins and intrinsic disordered proteins. Thirdly, the pre-trained models that integrate knowledge such as gene ontology are described. Fourthly, we briefly introduce pre-trained models in RNA fields. Finally, we introduce the most recent developments in protein designs and discuss the relationship of these models with the aforementioned pre-trained models that integrate protein structure information.
-
Keywords:
- protein foundation model /
- protein multi-modal model /
- protein structure /
- machine learning
[1] Senior A W, Evans R, Jumper J, Kirkpatrick J, Sifre L, Green T, Qin C, Žídek A, Nelson A W, Bridgland A, Penedones H, Petersen S, Simonyan K, Crossan S, Kohli P, Jones D T, Silver D, Kavukcuoglu K, Hassabis D 2020 Nature 577 706
 Google Scholar
Google Scholar
[2] Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Žídek A, Potapenko A, Bridgland A, Meyer C, Kohl S A A, Ballard A J, Cowie A, Romera-Paredes B, Nikolov S, Jain R, Adler J, Back T, Petersen S, Reiman D, Clancy E, Zielinski M, Steinegger M, Pacholska M, Berghammer T, Bodenstein S, Silver D, Vinyals O, Senior A W, Kavukcuoglu K, Kohli P, Hassabis D 2021 Nature 596 583
 Google Scholar
Google Scholar
[3] Radford A, Narasimhan K, Salimans T, Sutskever I 2018 Improving Language Understanding by Generative Pre-Training [2024-6-9]
[4] Radford A, Wu J, Child R, Luan D, Amodei D, Sutskever I 2019 Language Models are Unsupervised Multitask Learners [2024-6-9]
[5] Brown T B, Mann B, Ryder N, Subbiah M, Kaplan J, Dhariwal P, Neelakantan A, Shyam P, Sastry G, Askell A, Agarwal S, Herbert-Voss A, Krueger G, Henighan T, Child R, Ramesh A, Ziegler D M, Wu J, Winter C, Hesse C, Chen M, Sigler E, Litwin M, Gray S, Chess B, Clark J, Berner C, McCandlish S, Radford A, Sutskever I, Amodeis D 2020 arXiv: 2005.14165[cs.CV]
[6] Ouyang L, Wu J, Jiang X, Almeida D, Wainwright C L, Mishkin P, Zhang C, Agarwal S, Slama K, Ray A, Schulman J, Hilton J, Kelton F, Miller L, Simens M, Askell A, Welinder P, Christiano P, Leike J, Low R 2022 arXiv: 2203.02155[cs.CV]
[7] Devlin J, Chang M W, Lee K, Toutanova K 2018 arXiv: 1810.04805[cs.CV]
[8] Ma Z, He J, Qiu J, Cao H, Wang Y, Sun Z, Zheng L, Wang H, Tang S, Zheng T, Lin J, Feng G, Huang Z, Gao J, Zeng A, Zhang J, Zhong R, Shi T, Liu S, Zheng W, Tang J, Yang H, Liu X, Zhai J, Chen W 2022 Proceedings of the 27th ACM SIGPLAN Symposium on Principles and Practice of Parallel Programming Seoul, Republic of Korea, April 2–6, 2022 p192
[9] Han X, Zhang Z, Ding N, Gu Y, Liu X, Huo Y, Qiu J, Yao Y, Zhang A, Zhang L, Han W, Huang M, Jin Q, Lan Y, Liu Y, Liu Z, Lu Z, Qiu X, Song R, Tang J, Wen J R, Yuan J, Zhao W X, Zhu J 2021 arXiv: 2106.07139[AI]
[10] Yuan S, Zhao H, Zhao S, et al. 2022 arXiv: 2203.14101 [cs.LG]
[11] Wei J, Tay Y, Bommasani R, Raffel C, Zoph B, Borgeaud S, Yogatama D, Bosma M, Zhou D, Metzler D, Chi E H, Hashimoto T, Vinyals O, Liang P, Dean J, Fedus W 2022 arXiv: 2206.07682[cs.CV]
[12] Alayrac J B, Donahue J, Luc P, Miech A, Barr I, Hasson Y, Lenc K, Mensch A, Millican K, Reynolds M, Ring R, Rutherford E, Cabi S, Han T, Gong Z, Samangooei S, Monteiro M, Menick J, Borgeaud S, Brock A, Nematzadeh A, Sharifzadeh S, Binkowski M, Barreira R, Vinyals O, Zisserman A, Simonyan K 2022 arXiv: 2204.14198[cs.CV]
[13] OpenAI, Achiam J, Adler S, et al. 2024 arXiv: 2303.08774 [cs.CV]
[14] Driess D, Xia F, Sajjadi M S M, Lynch C, Chowdhery A, Ichter B, Wahid A, Tompson J, Vuong Q, Yu T, Huang W, Chebotar Y, Sermanet P, Duckworth D, Levine S, Vanhoucke V, Hausman K, Toussaint M, Greff K, Zeng A, Mordatch I, Florence P 2023 arXiv: 2303.03378[cs.LG]
[15] Touvron H, Lavril T, Izacard G, Martinet X, Lachaux M A, Lacroix T, Rozière B, Goyal N, Hambro E, Azhar F, Rodriguez A, Joulin A, Grave E, Lample G 2023 arXiv: 2302.13971[cs.CV]
[16] Gemini Team Google, Anil R, Borgeaud S, et al. 2024 arXiv: 2312.11805[cs.CV]
[17] Chen F, Han M, Zhao H, Zhang Q, Shi J, Xu S, Xu B 2023 arXiv: 2305.04160[cs.CV]
[18] Li K, He Y, Wang Y, Li Y, Wang W, Luo P, Wang Y, Wang L, Qiao Y 2023 arXiv: 2305.06355[cs.CV]
[19] Bepler T, Berger B 2019 arXiv: 1902.08661[cs.LG]
[20] Heinzinger M, Elnaggar A, Wang Y, Dallago C, Nechaev D, Matthes F, Rost B 2019 bioRxiv: 614313[Bioinformatics]
[21] Alley E C, Khimulya G, Biswas S, Alquraishi M, Church G M 2019 Nat. Methods 16 1315
 Google Scholar
Google Scholar
[22] Rives A, Meier J, Sercu T, Goyal S, Lin Z, Liu J, Guo D, Ott M, Zitnick C L, Ma J, Fergus R 2021 Proc. Natl. Acad. Sci. 118 e2016239118
 Google Scholar
Google Scholar
[23] Rao R, Liu J, Verkuil R, et al. 2021 bioRxiv: 2021.02.12. 430858 [Synthetic Biology]
[24] Meier J, Rao R, Verkuil R, Liu J, Sercu T, Rives A 2021 Advances in Neural Information Processing Systems 34 29287
 Google Scholar
Google Scholar
[25] Lin Z, Akin H, Rao R, Hie B, Zhu Z, Lu W, Smetanin N, Verkuil R, Kabeli O, Shmueli Y, dos Santos Costa A, Fazel-Zarandi M, Sercu T, Candido S, Rives A 2023 Science 379 1123
 Google Scholar
Google Scholar
[26] Lin Z, Akin H, Rao R, Hie B, Zhu Z, Lu W, Santos Costa A d, Fazel-Zarandi M, Sercu T, Candido S, Rives A 2022 bioRxiv: 2022.07.20.500902[Synthetic Biology]
[27] Madani A, McCann B, Naik N, Keskar N S, Anand N, Eguchi R R, Huang P S, Socher R 2020 arXiv: 2004.03497[q-bio.QM]
[28] Madani A, Krause B, Greene E R, Subramanian S, Mohr B P, Holton J M, Olmos J L, Xiong C, Sun Z Z, Socher R, Fraser J S, Naik N 2023 Nat. Biotechnol. 41 1099
 Google Scholar
Google Scholar
[29] He L, Zhang S, Wu L, Xia H, Ju F, Zhang H, Liu S, Xia Y, Zhu J, Deng P, Shao B, Qin T, Liu T Y 2021 arXiv: 2110.15527[cs.CV]
[30] Elnaggar A, Heinzinger M, Dallago C, Rihawi G, Wang Y, Jones L, Gibbs T, Feher T, Angerer C, Steinegger M, Bhowmik D, Rost B 2021 arXiv: 2007.06225[cs.LG]
[31] Chen B, Cheng X, Li P, Geng Y, Gong J, Li S, Bei Z, Tan X, Wang B, Zeng X, Liu C, Zeng A, Dong Y, Tang J, Song L 2024 arXiv: 2401.06199[q-bio.QM]
[32] Nguyen E, Poli M, Durrant M G, Thomas A W, Kang B, Sullivan J, Ng M Y, Lewis A, Patel A, Lou A, Ermon S, Baccus S A, Hernandez-Boussard T, Ré C, Hsu P D, Hie B L 2024 bioRxiv: 2024.02.27.582234[Synthetic Biology]
[33] Gao W, Mahajan S P, Sulam J, Gray J J 2020 Patterns 1 100142
 Google Scholar
Google Scholar
[34] Unsal S, Atas H, Albayrak M, Turhan K, Acar A C, Doğan T 2022 Nature Machine Intelligence 4 227
 Google Scholar
Google Scholar
[35] Zhang Q, Ding K, Lyv T, Wang X, Yin Q, Zhang Y, Yu J, Wang Y, Li X, Xiang Z, Feng K, Zhuang X, Wang Z, Qin M, Zhang M, Zhang J, Cui J, Huang T, Yan P, Xu R, Chen H, Li X, Fan X, Xing H, Chen H 2024 arXiv: 2401. 14656[cs.CV]
[36] 管星悦, 黄恒焱, 彭华祺, 刘彦航, 李文飞, 王炜 2023 物理学报 72 248708
 Google Scholar
Google Scholar
Guan X Y, Huang H Y, Peng H Q, Liu Y H, Li W F, Wang W 2023 Acta Phys. Sin. 72 248708
 Google Scholar
Google Scholar
[37] 陈光临, 张志勇 2023 物理学报 72 248705
 Google Scholar
Google Scholar
Chen G L, Zhang Z Y 2023 Acta Phys. Sin. 72 248705
 Google Scholar
Google Scholar
[38] 张嘉晖 2024 物理学报 73 069301
 Google Scholar
Google Scholar
Zhang J H 2024 Acta Phys. Sin. 73 069301
 Google Scholar
Google Scholar
[39] Zeng C, Jian Y, Vosoughi S, Zeng C, Zhao Y 2023 Nat. Commun. 14 1060
 Google Scholar
Google Scholar
[40] Zeng C, Zhao Y 2023 Scientia Sinica Physica, Mechanica & Astronomica 53 290018
 Google Scholar
Google Scholar
[41] Huh M, Cheung B, Wang T, Isola P 2024 arXiv: 2405.07987 [cs.LG]
[42] Bepler T, Berger B 2021 Cell Systems 12 654
 Google Scholar
Google Scholar
[43] Guo Y, Wu J, Ma H, Huang J 2022 Proceedings of the AAAI Conference on Artificial Intelligence 36 6801
 Google Scholar
Google Scholar
[44] Hermosilla P, Ropinski T 2022 arXiv: 2205.15675[q-bio.BM]
[45] Zhang Z, Xu M, Jamasb A, Chenthamarakshan V, Lozano A, Das P, Tang J 2022 arXiv: 2203.06125[cs.LG]
[46] Zhang Z, Xu M, Lozano A, Chenthamarakshan V, Das P, Tang J 2023 arXiv: 2303.06275[q-bio.QM]
[47] Gligorijević V, Renfrew P D, Kosciolek T, Leman J K, Berenberg D, Vatanen T, Chandler C, Taylor B C, Fisk I M, Vlamakis H, Xavier R J, Knight R, Cho K, Bonneau R 2021 Nat. Commun. 12 3168
 Google Scholar
Google Scholar
[48] Wang Z, Combs S A, Brand R, Calvo M R, Xu P, Price G, Golovach N, Salawu E O, Wise C J, Ponnapalli S P, Clark P M 2022 Sci. Rep. 12 6832
 Google Scholar
Google Scholar
[49] Chen C, Zhou J, Wang F, Liu X, Dou D 2023 arXiv: 2204.04213[cs.LG]
[50] Zhou G, Gao Z, Ding Q, Zheng H, Xu H, Wei Z, Zhang L, Ke G 2022 DOI: 10.26434/chemrxiv-2022-jjm0j-v4
[51] Su J, Han C, Zhou Y, Shan J, Zhou X, Yuan F 2023 bioRxiv: 2023.10.01.560349[Bioinformatics]
[52] Su J, Li Z, Han C, Zhou Y, Shan J, Zhou X, Ma D, OPMC T, Ovchinnikov S, Yuan F 2024 bioRxiv: 2024.05.24.595648[Bioinformatics]
[53] Hu M Y, Yuan F J, Yang K K, Ju F S, Su J, Wang H, Yang F, Ding Q Y 2022 arXiv:2206.06583 [q-bio.QM]
[54] Abramson J, Adler J, Dunger J, et al. 2024 Nature 630 493
 Google Scholar
Google Scholar
[55] Wang L, Liu H, Liu Y, Kurtin J, Ji S 2022 arXiv: 2207.12600[cs.LG]
[56] Somnath V R, Bunne C, Krause A 2021 arXiv: 2204.02337[cs.LG]
[57] Gainza P, Sverrisson F, Monti F, Rodola E, Boscaini D, Bronstein M M, Correia B E 2020 Nat. Methods 17 184
 Google Scholar
Google Scholar
[58] Wu F, Jin S, Jiang Y, Jin X, Tang B, Niu Z, Liu X, Zhang Q, Zeng X, Li S Z 2022 arXiv: 2204.08663[CE]
[59] Meyer T, D'Abramo M, Rueda M, Ferrer-Costa C, Pérez A, Carrillo O, Camps J, Fenollosa C, Repchevsky D, Gelpí J L, Orozco M 2010 Structure 18 1399
 Google Scholar
Google Scholar
[60] Zhang N, Bi Z, Liang X, Cheng S, Hong H, Deng S, Lian J, Zhang Q, Chen H 2022 arXiv: 2201.11147[q-bio.BM]
[61] Gu Y, Tinn R, Cheng H, Lucas M, Usuyama N, Liu X, Naumann T, Gao J, Poon H 2021 arXiv: 2007.15779[cs.CV]
[62] Zhou H Y, Fu Y, Zhang Z, Bian C, Yu Y 2023 arXiv: 2301.13154[cs.LG]
[63] Xu M, Yuan X, Miret S, Tang J 2023 arXiv: 2301.12040 [q-bio.BM]
[64] Singh J, Hanson J, Paliwal K, Zhou Y 2019 Nat. Commun. 10 5407
 Google Scholar
Google Scholar
[65] Singh J, Paliwal K, Zhang T, Singh J, Litfin T, Zhou Y 2021 Bioinformatics 37 2589
 Google Scholar
Google Scholar
[66] Wang J, Mao K, Zhao Y, Zeng C, Xiang J, Zhang Y, Xiao Y 2017 Nucleic Acids Res. 45 6299
 Google Scholar
Google Scholar
[67] Wang J, Xiao Y 2017 Current Protocols in Bioinformatics 57 5
 Google Scholar
Google Scholar
[68] Wang J, Wang J, Huang Y, Xiao Y 2019 Int. J. Mol. Sci. 20 4116
 Google Scholar
Google Scholar
[69] Tan Y L, Wang X, Shi Y Z, Zhang W, Tan Z J 2022 Biophys. J. 121 142
 Google Scholar
Google Scholar
[70] Zhou L, Wang X, Yu S, Tan Y L, Tan Z J 2022 Biophys. J. 121 3381
 Google Scholar
Google Scholar
[71] Wang X, Tan Y L, Yu S, Shi Y Z, Tan Z J 2023 Biophys. J. 122 1503
 Google Scholar
Google Scholar
[72] Li J, Zhu W, Wang J, Li W, Gong S, Zhang J, Wang W 2018 PLoS Comput. Biol. 14 e1006514
 Google Scholar
Google Scholar
[73] Fu L, Cao Y, Wu J, Peng Q, Nie Q, Xie X 2022 Nucleic Acids Res. 50 e14
 Google Scholar
Google Scholar
[74] Pearce R, Omenn G S, Zhang Y 2022 bioRxiv: 2022. 05.15.491755[Bioinformatics]
[75] Baek M, McHugh R, Anishchenko I, Baker D, DiMaio F 2022 bioRxiv: 2022.09.09.507333[Bioinformatics]
[76] Zhang J, Lang M, Zhou Y, Zhang Y 2024 Trends in Genetics 40 94
 Google Scholar
Google Scholar
[77] Li J, Zhou Y, Chen S J 2024 Curr. Opin. Struct. Biol. 87 102847
 Google Scholar
Google Scholar
[78] Chen J, Hu Z, Sun S, Tan Q, Wang Y, Yu Q, Zong L, Hong L, Xiao J, Shen T, King I, Li Y 2022 arXiv: 2204.00300[q-bio.QM]
[79] Chen K, Zhou Y, Ding M, Wang Y, Ren Z, Yang Y 2023 bioRxiv: 2023.01.31.526427[Bioinformatics]
[80] Babjac A N, Lu Z, Emrich S J 2023 Proceedings of the 14th ACM International Conference on Bioinformatics, Computational Biology, and Health Informatics New York, United States, September 3–6, 2023 p1
[81] Chu Y, Yu D, Li Y, Huang K, Shen Y, Cong L, Zhang J, Wang M 2024 Nature Machine Intelligence 6 449
 Google Scholar
Google Scholar
[82] Yang Y, Li G, Pang K, Cao W, Li X, Zhang Z 2023 bioRxiv: 2023.09.08.556883[Bioinformatics]
[83] Zhang Y, Lang M, Jiang J, Gao Z, Xu F, Litfin T, Chen K, Singh J, Huang X, Song G, Tian Y, Zhan J, Chen J, Zhou Y 2024 Nucleic Acids Res. 52 e3
 Google Scholar
Google Scholar
[84] Wang X, Gu R, Chen Z, Li Y, Ji X, Ke G, Wen H 2023 bioRxiv: 2023.07.11.548588[Bioinformatics]
[85] Wang N, Bian J, Li Y, Li X, Mumtaz S, Kong L, Xiong H 2024 Nature Machine Intelligence 6 548
 Google Scholar
Google Scholar
[86] Akiyama M, Sakakibara Y 2022 NAR Genomics and Bioinformatics 4 lqac012
 Google Scholar
Google Scholar
[87] Shen T, Hu Z, Peng Z, Chen J, Xiong P, Hong L, Zheng L, Wang Y, King I, Wang S, Siqi S, Yu L 2022 arXiv: 2207.01586[q-bio.QM]
[88] Li Y, Zhang C, Feng C, Pearce R, Lydia Freddolino P, Zhang Y 2023 Nat. Commun. 14 5745
 Google Scholar
Google Scholar
[89] Ferruz N, Schmidt S, Höcker B 2022 Nat. Commun. 13 4348
 Google Scholar
Google Scholar
[90] Wang J, Lisanza S, Juergens D, Tischer D, Watson J L, Castro K M, Ragotte R, Saragovi A, Milles L F, Baek M, Anishchenko I, Yang W, Hicks D R, Expòsit M, Schlichthaerle T, Chun J H, Dauparas J, Bennett N, Wicky B I M, Muenks A, DiMaio F, Correia B, Ovchinnikov S, Baker D 2022 Science 377 387
 Google Scholar
Google Scholar
[91] Trippe B L, Yim J, Tischer D, Baker D, Broderick T, Barzilay R, Jaakkola T 2022 arXiv: 2206.04119[q-bio.BM]
[92] Anishchenko I, Pellock S J, Chidyausiku T M, Ramelot T A, Ovchinnikov S, Hao J, Bafna K, Norn C, Kang A, Bera A K, DiMaio F, Carter L, Chow C M, Montelione G T, Baker D 2021 Nature 600 547
 Google Scholar
Google Scholar
[93] Wicky B I M, Milles L F, Courbet A, Ragotte R J, Dauparas J, Kinfu E, Tipps S, Kibler R D, Baek M, DiMaio F, Li X, Carter L, Kang A, Nguyen H, Bera A K, Baker D 2022 Science 378 56
 Google Scholar
Google Scholar
[94] Anand N, Achim T 2022 arXiv: 2205.15019[q-bio.QM]
[95] Luo S, Su Y, Peng X, Wang S, Peng J, Ma J 2022 Advances in Neural Information Processing Systems 35 9754
 Google Scholar
Google Scholar
[96] Cao L, Coventry B, Goreshnik I, et al 2022 Nature 605 551
 Google Scholar
Google Scholar
[97] Kuhlman B, Bradley P 2019 Nat. Rev. Mol. Cell Biol. 20 681
 Google Scholar
Google Scholar
[98] Pan X, Kortemme T 2021 J. Biol. Chem. 296 100558
 Google Scholar
Google Scholar
[99] Khakzad H, Igashov I, Schneuing A, Goverde C, Bronstein M, Correia B 2023 Cell Systems 14 925
 Google Scholar
Google Scholar
[100] Malbranke C, Bikard D, Cocco S, Monasson R, Tubiana J 2023 Curr. Opin. Struct. Biol. 80 102571
 Google Scholar
Google Scholar
[101] Kortemme T 2024 Cell 187 526
 Google Scholar
Google Scholar
[102] Notin P, Rollins N, Gal Y, Sander C, Marks D 2024 Nat. Biotechnol. 42 216
 Google Scholar
Google Scholar
[103] Listov D, Goverde C A, Correia B E, Fleishman S J 2024 Nat. Rev. Mol. Cell Biol. 25 639
 Google Scholar
Google Scholar
[104] Ingraham J, Garg V K, Barzilay R, Jaakkola T 2019 Proceedings of the 33rd International Conference on Neural Information Processing Systems Vancouver, BC, Canada, December 8–14, 2019 p15820
[105] Dauparas J, Anishchenko I, Bennett N, Bai H, Ragotte R J, Milles L F, Wicky B I M, Courbet A, de Haas R J, Bethel N, Leung P J Y, Huddy T F, Pellock S, Tischer D, Chan F, Koepnick B, Nguyen H, Kang A, Sankaran B, Bera A K, King N P, Baker D 2022 Science 378 49
 Google Scholar
Google Scholar
[106] Hsu C, Verkuil R, Liu J, Lin Z, Hie B, Sercu T, Lerer A, Rives A 2022 bioRxiv: 2022.04.10.487779[Systems Biology]
[107] Sohl-Dickstein J, Weiss E A, Maheswaranathan N, Ganguli S 2015 arXiv: 1503.03585[cs.LG]
[108] Ho J, Jain A, Abbeel P 2020 Advances in Neural Information Processing Systems 33 6840
 Google Scholar
Google Scholar
[109] Watson J L, Juergens D, Bennett N R, et al 2023 Nature 620 1089
 Google Scholar
Google Scholar
[110] Song Y, Sohl-Dickstein J, Kingma D P, Kumar A, Ermon S, Poole B 2020 arXiv: 2011.13456[cs.LG]
[111] Lee J S, Kim J, Kim P M 2023 Nature Computational Science 3 382
 Google Scholar
Google Scholar
[112] Liu Y, Chen L, Liu H 2023 bioRxiv: 2023.11.18.567666 [Bioinformatics]
[113] Zheng Z, Deng Y, Xue D, Zhou Y, YE F, Gu Q 2023 arXiv: 2302.01649[cs.LG]
[114] Yang K K, Zanichelli N, Yeh H 2023 Protein Eng. Des. Sel. 36 gzad015
 Google Scholar
Google Scholar
[115] Kaplan J, McCandlish S, Henighan T, Brown T B, Chess B, Child R, Gray S, Radford A, Wu J, Amodei D 2020 arXiv: 2001.08361[cs.LG]
[116] He K, Chen X, Xie S, Li Y, Dollár P, Girshick R 2021 arXiv: 2111.06377[cs.CV]
[117] Chen T, Kornblith S, Norouzi M, Hinton G 2020 arXiv: 2002.05709[cs.LG]
[118] Wang Z, Wang Z, Srinivasan B, Ioannidis V N, Rangwala H, Anubhai R 2023 arXiv: 2310.03320[cs.LG]
[119] Von Rueden L, Mayer S, Beckh K, Georgiev B, Giesselbach S, Heese R, Kirsch B, Walczak M, Pfrommer J, Pick A, Ramamurthy R, Garcke J, Bauckhage C, Schuecker J 2021 IEEE Trans. Knowl. Data Eng. 35 614
[120] Bao L, Zhang X, Jin L, Tan Z J 2015 Chin. Phys. B 25 018703
 Google Scholar
Google Scholar
[121] Qiang X W, Zhang C, Dong H L, Tian F J, Fu H, Yang Y J, Dai L, Zhang X H, Tan Z J 2022 Phys. Rev. Lett. 128 108103
 Google Scholar
Google Scholar
[122] Dong H L, Zhang C, Dai L, Zhang Y, Zhang X H, Tan Z J 2024 Nucleic Acids Res. 52 2519
 Google Scholar
Google Scholar
-
表 1 多模态蛋白质预训练模型
Table 1. Multimodal protein pre-trained models.
模型名 时间 模型 数据模态 预训练方法 训练集 参数量 算力要求 下游任务 文献 融合了结构信息的通用蛋白质预训练模型 Bepler &Berger 2019 Bi-LSTM Sequence, structure MLM for sequences, supervised learning for 3D structures 76M sequences, 28K structures — 1X 32G-V100, 13 to 51 days Fold classification transmembrane region prediction [19,42] Guo model 2022 CNN Structure Self-supervised pre-training on noised pair-distance 73K structures — — QA, PPI [43] New IEConv 2022 GCN Sequence, structure Contrastive learning between randomly sampled 3D substructures 476K chains 30M — protein function prediction, protein fold classification, structural similarity prediction, protein-ligand binding affinity prediction [44] GearNet 2023 ESM-1b, GearNet Sequence, structure PLM, contrastive learning 805K structures from AlphaFoldDB — 4X A100 Fold classification, EC, GO STEPS 2023 BERT, GCN Sequence, structure PLM, supervised learning from 3D structures 40K structures — — Membrane protein classification, cellular location prediction, EC UNI-MOL 2023 Transformer Sequence, structure Atom 3D position denoise, masked atom type prediction 209M molecule conformations, 3.2M protein pockets structure — 8X 32G-V100, 3 days molecular property prediction, molecular conformation generation, pocket property prediction, protein-ligand binding pose prediction SaProt 2023 BERT Sequence, structure Convert structures to structure-aware vocabulary, MLM 40M sequences and structures from PDB/AlphaFoldDB 650M 64X 80G-A100, 3 months Thermostability, HumanPPI, Metal Ion Binding, EC, GO, DeepLoc, contact prediction [51] 融合了结构信息的非通用蛋白质预训练模型 Evoformer 2021 Evoformer Sequence, structure MLM, Supervised learning BPD+Uniclust30, PDB — 128TPU-v3, 11 days Structure prediction [2] DeepFRI 2021 LSTM+GCN Sequence, structure PLM(pretrained, frozen), supervised learning for 3D structures 10M sequences for pre-training — — GO, EC, PPI interaction sites [47] LM-GVP 2022 Transformer +GVP Sequence, structure PLM(changeable), supervised learning for 3D structures — — 8X 32G-V100 fluorescence, protease stability, GO, mutational effects [48] ProNet 2023 GCN Sequence, structure Supervised learning — — — Fold classification, reaction classification, binding affinity, PI HoloProt 2022 MPN Sequence, structure surface Supervised learning — 1.8M 1X 1080Ti, 1 day Ligand binding affinity, EC [56] 编码动态三维结构信息的预训练模型 ProtMD 2022 E(3)-Equivariant Graph Matching Network Sequence, structure trajectory Self-supervised learning, atom-level prompt-based denoising generative task, conformation-level snapshot ordering task 62.8K snapshots from MD for 64 protein-ligand pairs 5.2M 4X V100 Binding affinity prediction, binary classification of ligand efficacy [58] 融合了知识的蛋白质预训练模型 OntoProtein 2022 ProtBert, Gu-model Sequence, knowledge MLM, contrastive learning ProteinKG25 with 5M knowledge triples — V100 TAPE, PPI, Protein function prediction [60] KeAP 2023 ProtBert, Gu-model Sequence, knowledge MLM ProteinKG25 — — TAPE, PPI, Protein function prediction [62] ProtST 2023 ProtBert, ESM-1b,
ESM-2, PubMedBertSequence, knowledge MLM, Multimodal Representation Alignment, Multimodal Mask Prediction ProtDescribe with 553K sequence-property pairs — 4X V100 Protein localization prediction, Fitness landscape prediction, Protein function annotation [63] RNA语言模型 RNA-FM 2022.8 BERT Sequence MLM RNAcentral, 23.7M ncRNA sequences — 8X A100 80G, 1 month SS prediction, 3D contact/distance map, 3D reconstruction, evolution study, RNA-protein interaction, MRL prediction [78] RNABert 2022 BERT Sequence MLM RNAcentral (762K) & Rfam 14.3 dataset — V100 structural alignment, clustering [86] SpliceBERT 2023 BERT Sequence MLM Pre-mRNA of 72 vertebrates, 2M sequences, 64B nucleotides 19.4M 8X V100, 1 week multi-species splice site prediction, human branch point prediction [79] RNA-MSM 2023 MSA-transformer Sequence MLM 4069 RNA families from Rfam 14.7 — 8X V100 32G SS prediction, solvent accessibility prediction [83] Uni-RNA 2023 BERT Sequence MLM RNAcentral & nt & GWH (1billion sequences) 25—400M 128X A100 SS prediction, 3D structure prediction, MRL, Isoform percentage prediction on 3’UTR, splice site prediction, classification of ncRNA functional families, modification site prediction [84] RNAErnie 2024 ERNIE Sequence, motif information MLM at base/subsequence/motif level masking RNAcentral, 23M ncRNA sequences 105M 4X V100 32G, 250 hours sequence classification, RNA–RNA interaction, SS prediction [85] *PLM, protein language model; MLM, masked language model; GCN, graph convolutional network; GVP, geometric vector perceptrons; EC, enzyme commission number prediction; GO, gene ontology term prediction; PPI, protein-protein interaction; TAPE, the tasks assessing protein embeddings database; QA, quality assessment of structures; SS, secondary structure; MRL, mean ribosome load prediction in mRNA. -
[1] Senior A W, Evans R, Jumper J, Kirkpatrick J, Sifre L, Green T, Qin C, Žídek A, Nelson A W, Bridgland A, Penedones H, Petersen S, Simonyan K, Crossan S, Kohli P, Jones D T, Silver D, Kavukcuoglu K, Hassabis D 2020 Nature 577 706
 Google Scholar
Google Scholar
[2] Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Žídek A, Potapenko A, Bridgland A, Meyer C, Kohl S A A, Ballard A J, Cowie A, Romera-Paredes B, Nikolov S, Jain R, Adler J, Back T, Petersen S, Reiman D, Clancy E, Zielinski M, Steinegger M, Pacholska M, Berghammer T, Bodenstein S, Silver D, Vinyals O, Senior A W, Kavukcuoglu K, Kohli P, Hassabis D 2021 Nature 596 583
 Google Scholar
Google Scholar
[3] Radford A, Narasimhan K, Salimans T, Sutskever I 2018 Improving Language Understanding by Generative Pre-Training [2024-6-9]
[4] Radford A, Wu J, Child R, Luan D, Amodei D, Sutskever I 2019 Language Models are Unsupervised Multitask Learners [2024-6-9]
[5] Brown T B, Mann B, Ryder N, Subbiah M, Kaplan J, Dhariwal P, Neelakantan A, Shyam P, Sastry G, Askell A, Agarwal S, Herbert-Voss A, Krueger G, Henighan T, Child R, Ramesh A, Ziegler D M, Wu J, Winter C, Hesse C, Chen M, Sigler E, Litwin M, Gray S, Chess B, Clark J, Berner C, McCandlish S, Radford A, Sutskever I, Amodeis D 2020 arXiv: 2005.14165[cs.CV]
[6] Ouyang L, Wu J, Jiang X, Almeida D, Wainwright C L, Mishkin P, Zhang C, Agarwal S, Slama K, Ray A, Schulman J, Hilton J, Kelton F, Miller L, Simens M, Askell A, Welinder P, Christiano P, Leike J, Low R 2022 arXiv: 2203.02155[cs.CV]
[7] Devlin J, Chang M W, Lee K, Toutanova K 2018 arXiv: 1810.04805[cs.CV]
[8] Ma Z, He J, Qiu J, Cao H, Wang Y, Sun Z, Zheng L, Wang H, Tang S, Zheng T, Lin J, Feng G, Huang Z, Gao J, Zeng A, Zhang J, Zhong R, Shi T, Liu S, Zheng W, Tang J, Yang H, Liu X, Zhai J, Chen W 2022 Proceedings of the 27th ACM SIGPLAN Symposium on Principles and Practice of Parallel Programming Seoul, Republic of Korea, April 2–6, 2022 p192
[9] Han X, Zhang Z, Ding N, Gu Y, Liu X, Huo Y, Qiu J, Yao Y, Zhang A, Zhang L, Han W, Huang M, Jin Q, Lan Y, Liu Y, Liu Z, Lu Z, Qiu X, Song R, Tang J, Wen J R, Yuan J, Zhao W X, Zhu J 2021 arXiv: 2106.07139[AI]
[10] Yuan S, Zhao H, Zhao S, et al. 2022 arXiv: 2203.14101 [cs.LG]
[11] Wei J, Tay Y, Bommasani R, Raffel C, Zoph B, Borgeaud S, Yogatama D, Bosma M, Zhou D, Metzler D, Chi E H, Hashimoto T, Vinyals O, Liang P, Dean J, Fedus W 2022 arXiv: 2206.07682[cs.CV]
[12] Alayrac J B, Donahue J, Luc P, Miech A, Barr I, Hasson Y, Lenc K, Mensch A, Millican K, Reynolds M, Ring R, Rutherford E, Cabi S, Han T, Gong Z, Samangooei S, Monteiro M, Menick J, Borgeaud S, Brock A, Nematzadeh A, Sharifzadeh S, Binkowski M, Barreira R, Vinyals O, Zisserman A, Simonyan K 2022 arXiv: 2204.14198[cs.CV]
[13] OpenAI, Achiam J, Adler S, et al. 2024 arXiv: 2303.08774 [cs.CV]
[14] Driess D, Xia F, Sajjadi M S M, Lynch C, Chowdhery A, Ichter B, Wahid A, Tompson J, Vuong Q, Yu T, Huang W, Chebotar Y, Sermanet P, Duckworth D, Levine S, Vanhoucke V, Hausman K, Toussaint M, Greff K, Zeng A, Mordatch I, Florence P 2023 arXiv: 2303.03378[cs.LG]
[15] Touvron H, Lavril T, Izacard G, Martinet X, Lachaux M A, Lacroix T, Rozière B, Goyal N, Hambro E, Azhar F, Rodriguez A, Joulin A, Grave E, Lample G 2023 arXiv: 2302.13971[cs.CV]
[16] Gemini Team Google, Anil R, Borgeaud S, et al. 2024 arXiv: 2312.11805[cs.CV]
[17] Chen F, Han M, Zhao H, Zhang Q, Shi J, Xu S, Xu B 2023 arXiv: 2305.04160[cs.CV]
[18] Li K, He Y, Wang Y, Li Y, Wang W, Luo P, Wang Y, Wang L, Qiao Y 2023 arXiv: 2305.06355[cs.CV]
[19] Bepler T, Berger B 2019 arXiv: 1902.08661[cs.LG]
[20] Heinzinger M, Elnaggar A, Wang Y, Dallago C, Nechaev D, Matthes F, Rost B 2019 bioRxiv: 614313[Bioinformatics]
[21] Alley E C, Khimulya G, Biswas S, Alquraishi M, Church G M 2019 Nat. Methods 16 1315
 Google Scholar
Google Scholar
[22] Rives A, Meier J, Sercu T, Goyal S, Lin Z, Liu J, Guo D, Ott M, Zitnick C L, Ma J, Fergus R 2021 Proc. Natl. Acad. Sci. 118 e2016239118
 Google Scholar
Google Scholar
[23] Rao R, Liu J, Verkuil R, et al. 2021 bioRxiv: 2021.02.12. 430858 [Synthetic Biology]
[24] Meier J, Rao R, Verkuil R, Liu J, Sercu T, Rives A 2021 Advances in Neural Information Processing Systems 34 29287
 Google Scholar
Google Scholar
[25] Lin Z, Akin H, Rao R, Hie B, Zhu Z, Lu W, Smetanin N, Verkuil R, Kabeli O, Shmueli Y, dos Santos Costa A, Fazel-Zarandi M, Sercu T, Candido S, Rives A 2023 Science 379 1123
 Google Scholar
Google Scholar
[26] Lin Z, Akin H, Rao R, Hie B, Zhu Z, Lu W, Santos Costa A d, Fazel-Zarandi M, Sercu T, Candido S, Rives A 2022 bioRxiv: 2022.07.20.500902[Synthetic Biology]
[27] Madani A, McCann B, Naik N, Keskar N S, Anand N, Eguchi R R, Huang P S, Socher R 2020 arXiv: 2004.03497[q-bio.QM]
[28] Madani A, Krause B, Greene E R, Subramanian S, Mohr B P, Holton J M, Olmos J L, Xiong C, Sun Z Z, Socher R, Fraser J S, Naik N 2023 Nat. Biotechnol. 41 1099
 Google Scholar
Google Scholar
[29] He L, Zhang S, Wu L, Xia H, Ju F, Zhang H, Liu S, Xia Y, Zhu J, Deng P, Shao B, Qin T, Liu T Y 2021 arXiv: 2110.15527[cs.CV]
[30] Elnaggar A, Heinzinger M, Dallago C, Rihawi G, Wang Y, Jones L, Gibbs T, Feher T, Angerer C, Steinegger M, Bhowmik D, Rost B 2021 arXiv: 2007.06225[cs.LG]
[31] Chen B, Cheng X, Li P, Geng Y, Gong J, Li S, Bei Z, Tan X, Wang B, Zeng X, Liu C, Zeng A, Dong Y, Tang J, Song L 2024 arXiv: 2401.06199[q-bio.QM]
[32] Nguyen E, Poli M, Durrant M G, Thomas A W, Kang B, Sullivan J, Ng M Y, Lewis A, Patel A, Lou A, Ermon S, Baccus S A, Hernandez-Boussard T, Ré C, Hsu P D, Hie B L 2024 bioRxiv: 2024.02.27.582234[Synthetic Biology]
[33] Gao W, Mahajan S P, Sulam J, Gray J J 2020 Patterns 1 100142
 Google Scholar
Google Scholar
[34] Unsal S, Atas H, Albayrak M, Turhan K, Acar A C, Doğan T 2022 Nature Machine Intelligence 4 227
 Google Scholar
Google Scholar
[35] Zhang Q, Ding K, Lyv T, Wang X, Yin Q, Zhang Y, Yu J, Wang Y, Li X, Xiang Z, Feng K, Zhuang X, Wang Z, Qin M, Zhang M, Zhang J, Cui J, Huang T, Yan P, Xu R, Chen H, Li X, Fan X, Xing H, Chen H 2024 arXiv: 2401. 14656[cs.CV]
[36] 管星悦, 黄恒焱, 彭华祺, 刘彦航, 李文飞, 王炜 2023 物理学报 72 248708
 Google Scholar
Google Scholar
Guan X Y, Huang H Y, Peng H Q, Liu Y H, Li W F, Wang W 2023 Acta Phys. Sin. 72 248708
 Google Scholar
Google Scholar
[37] 陈光临, 张志勇 2023 物理学报 72 248705
 Google Scholar
Google Scholar
Chen G L, Zhang Z Y 2023 Acta Phys. Sin. 72 248705
 Google Scholar
Google Scholar
[38] 张嘉晖 2024 物理学报 73 069301
 Google Scholar
Google Scholar
Zhang J H 2024 Acta Phys. Sin. 73 069301
 Google Scholar
Google Scholar
[39] Zeng C, Jian Y, Vosoughi S, Zeng C, Zhao Y 2023 Nat. Commun. 14 1060
 Google Scholar
Google Scholar
[40] Zeng C, Zhao Y 2023 Scientia Sinica Physica, Mechanica & Astronomica 53 290018
 Google Scholar
Google Scholar
[41] Huh M, Cheung B, Wang T, Isola P 2024 arXiv: 2405.07987 [cs.LG]
[42] Bepler T, Berger B 2021 Cell Systems 12 654
 Google Scholar
Google Scholar
[43] Guo Y, Wu J, Ma H, Huang J 2022 Proceedings of the AAAI Conference on Artificial Intelligence 36 6801
 Google Scholar
Google Scholar
[44] Hermosilla P, Ropinski T 2022 arXiv: 2205.15675[q-bio.BM]
[45] Zhang Z, Xu M, Jamasb A, Chenthamarakshan V, Lozano A, Das P, Tang J 2022 arXiv: 2203.06125[cs.LG]
[46] Zhang Z, Xu M, Lozano A, Chenthamarakshan V, Das P, Tang J 2023 arXiv: 2303.06275[q-bio.QM]
[47] Gligorijević V, Renfrew P D, Kosciolek T, Leman J K, Berenberg D, Vatanen T, Chandler C, Taylor B C, Fisk I M, Vlamakis H, Xavier R J, Knight R, Cho K, Bonneau R 2021 Nat. Commun. 12 3168
 Google Scholar
Google Scholar
[48] Wang Z, Combs S A, Brand R, Calvo M R, Xu P, Price G, Golovach N, Salawu E O, Wise C J, Ponnapalli S P, Clark P M 2022 Sci. Rep. 12 6832
 Google Scholar
Google Scholar
[49] Chen C, Zhou J, Wang F, Liu X, Dou D 2023 arXiv: 2204.04213[cs.LG]
[50] Zhou G, Gao Z, Ding Q, Zheng H, Xu H, Wei Z, Zhang L, Ke G 2022 DOI: 10.26434/chemrxiv-2022-jjm0j-v4
[51] Su J, Han C, Zhou Y, Shan J, Zhou X, Yuan F 2023 bioRxiv: 2023.10.01.560349[Bioinformatics]
[52] Su J, Li Z, Han C, Zhou Y, Shan J, Zhou X, Ma D, OPMC T, Ovchinnikov S, Yuan F 2024 bioRxiv: 2024.05.24.595648[Bioinformatics]
[53] Hu M Y, Yuan F J, Yang K K, Ju F S, Su J, Wang H, Yang F, Ding Q Y 2022 arXiv:2206.06583 [q-bio.QM]
[54] Abramson J, Adler J, Dunger J, et al. 2024 Nature 630 493
 Google Scholar
Google Scholar
[55] Wang L, Liu H, Liu Y, Kurtin J, Ji S 2022 arXiv: 2207.12600[cs.LG]
[56] Somnath V R, Bunne C, Krause A 2021 arXiv: 2204.02337[cs.LG]
[57] Gainza P, Sverrisson F, Monti F, Rodola E, Boscaini D, Bronstein M M, Correia B E 2020 Nat. Methods 17 184
 Google Scholar
Google Scholar
[58] Wu F, Jin S, Jiang Y, Jin X, Tang B, Niu Z, Liu X, Zhang Q, Zeng X, Li S Z 2022 arXiv: 2204.08663[CE]
[59] Meyer T, D'Abramo M, Rueda M, Ferrer-Costa C, Pérez A, Carrillo O, Camps J, Fenollosa C, Repchevsky D, Gelpí J L, Orozco M 2010 Structure 18 1399
 Google Scholar
Google Scholar
[60] Zhang N, Bi Z, Liang X, Cheng S, Hong H, Deng S, Lian J, Zhang Q, Chen H 2022 arXiv: 2201.11147[q-bio.BM]
[61] Gu Y, Tinn R, Cheng H, Lucas M, Usuyama N, Liu X, Naumann T, Gao J, Poon H 2021 arXiv: 2007.15779[cs.CV]
[62] Zhou H Y, Fu Y, Zhang Z, Bian C, Yu Y 2023 arXiv: 2301.13154[cs.LG]
[63] Xu M, Yuan X, Miret S, Tang J 2023 arXiv: 2301.12040 [q-bio.BM]
[64] Singh J, Hanson J, Paliwal K, Zhou Y 2019 Nat. Commun. 10 5407
 Google Scholar
Google Scholar
[65] Singh J, Paliwal K, Zhang T, Singh J, Litfin T, Zhou Y 2021 Bioinformatics 37 2589
 Google Scholar
Google Scholar
[66] Wang J, Mao K, Zhao Y, Zeng C, Xiang J, Zhang Y, Xiao Y 2017 Nucleic Acids Res. 45 6299
 Google Scholar
Google Scholar
[67] Wang J, Xiao Y 2017 Current Protocols in Bioinformatics 57 5
 Google Scholar
Google Scholar
[68] Wang J, Wang J, Huang Y, Xiao Y 2019 Int. J. Mol. Sci. 20 4116
 Google Scholar
Google Scholar
[69] Tan Y L, Wang X, Shi Y Z, Zhang W, Tan Z J 2022 Biophys. J. 121 142
 Google Scholar
Google Scholar
[70] Zhou L, Wang X, Yu S, Tan Y L, Tan Z J 2022 Biophys. J. 121 3381
 Google Scholar
Google Scholar
[71] Wang X, Tan Y L, Yu S, Shi Y Z, Tan Z J 2023 Biophys. J. 122 1503
 Google Scholar
Google Scholar
[72] Li J, Zhu W, Wang J, Li W, Gong S, Zhang J, Wang W 2018 PLoS Comput. Biol. 14 e1006514
 Google Scholar
Google Scholar
[73] Fu L, Cao Y, Wu J, Peng Q, Nie Q, Xie X 2022 Nucleic Acids Res. 50 e14
 Google Scholar
Google Scholar
[74] Pearce R, Omenn G S, Zhang Y 2022 bioRxiv: 2022. 05.15.491755[Bioinformatics]
[75] Baek M, McHugh R, Anishchenko I, Baker D, DiMaio F 2022 bioRxiv: 2022.09.09.507333[Bioinformatics]
[76] Zhang J, Lang M, Zhou Y, Zhang Y 2024 Trends in Genetics 40 94
 Google Scholar
Google Scholar
[77] Li J, Zhou Y, Chen S J 2024 Curr. Opin. Struct. Biol. 87 102847
 Google Scholar
Google Scholar
[78] Chen J, Hu Z, Sun S, Tan Q, Wang Y, Yu Q, Zong L, Hong L, Xiao J, Shen T, King I, Li Y 2022 arXiv: 2204.00300[q-bio.QM]
[79] Chen K, Zhou Y, Ding M, Wang Y, Ren Z, Yang Y 2023 bioRxiv: 2023.01.31.526427[Bioinformatics]
[80] Babjac A N, Lu Z, Emrich S J 2023 Proceedings of the 14th ACM International Conference on Bioinformatics, Computational Biology, and Health Informatics New York, United States, September 3–6, 2023 p1
[81] Chu Y, Yu D, Li Y, Huang K, Shen Y, Cong L, Zhang J, Wang M 2024 Nature Machine Intelligence 6 449
 Google Scholar
Google Scholar
[82] Yang Y, Li G, Pang K, Cao W, Li X, Zhang Z 2023 bioRxiv: 2023.09.08.556883[Bioinformatics]
[83] Zhang Y, Lang M, Jiang J, Gao Z, Xu F, Litfin T, Chen K, Singh J, Huang X, Song G, Tian Y, Zhan J, Chen J, Zhou Y 2024 Nucleic Acids Res. 52 e3
 Google Scholar
Google Scholar
[84] Wang X, Gu R, Chen Z, Li Y, Ji X, Ke G, Wen H 2023 bioRxiv: 2023.07.11.548588[Bioinformatics]
[85] Wang N, Bian J, Li Y, Li X, Mumtaz S, Kong L, Xiong H 2024 Nature Machine Intelligence 6 548
 Google Scholar
Google Scholar
[86] Akiyama M, Sakakibara Y 2022 NAR Genomics and Bioinformatics 4 lqac012
 Google Scholar
Google Scholar
[87] Shen T, Hu Z, Peng Z, Chen J, Xiong P, Hong L, Zheng L, Wang Y, King I, Wang S, Siqi S, Yu L 2022 arXiv: 2207.01586[q-bio.QM]
[88] Li Y, Zhang C, Feng C, Pearce R, Lydia Freddolino P, Zhang Y 2023 Nat. Commun. 14 5745
 Google Scholar
Google Scholar
[89] Ferruz N, Schmidt S, Höcker B 2022 Nat. Commun. 13 4348
 Google Scholar
Google Scholar
[90] Wang J, Lisanza S, Juergens D, Tischer D, Watson J L, Castro K M, Ragotte R, Saragovi A, Milles L F, Baek M, Anishchenko I, Yang W, Hicks D R, Expòsit M, Schlichthaerle T, Chun J H, Dauparas J, Bennett N, Wicky B I M, Muenks A, DiMaio F, Correia B, Ovchinnikov S, Baker D 2022 Science 377 387
 Google Scholar
Google Scholar
[91] Trippe B L, Yim J, Tischer D, Baker D, Broderick T, Barzilay R, Jaakkola T 2022 arXiv: 2206.04119[q-bio.BM]
[92] Anishchenko I, Pellock S J, Chidyausiku T M, Ramelot T A, Ovchinnikov S, Hao J, Bafna K, Norn C, Kang A, Bera A K, DiMaio F, Carter L, Chow C M, Montelione G T, Baker D 2021 Nature 600 547
 Google Scholar
Google Scholar
[93] Wicky B I M, Milles L F, Courbet A, Ragotte R J, Dauparas J, Kinfu E, Tipps S, Kibler R D, Baek M, DiMaio F, Li X, Carter L, Kang A, Nguyen H, Bera A K, Baker D 2022 Science 378 56
 Google Scholar
Google Scholar
[94] Anand N, Achim T 2022 arXiv: 2205.15019[q-bio.QM]
[95] Luo S, Su Y, Peng X, Wang S, Peng J, Ma J 2022 Advances in Neural Information Processing Systems 35 9754
 Google Scholar
Google Scholar
[96] Cao L, Coventry B, Goreshnik I, et al 2022 Nature 605 551
 Google Scholar
Google Scholar
[97] Kuhlman B, Bradley P 2019 Nat. Rev. Mol. Cell Biol. 20 681
 Google Scholar
Google Scholar
[98] Pan X, Kortemme T 2021 J. Biol. Chem. 296 100558
 Google Scholar
Google Scholar
[99] Khakzad H, Igashov I, Schneuing A, Goverde C, Bronstein M, Correia B 2023 Cell Systems 14 925
 Google Scholar
Google Scholar
[100] Malbranke C, Bikard D, Cocco S, Monasson R, Tubiana J 2023 Curr. Opin. Struct. Biol. 80 102571
 Google Scholar
Google Scholar
[101] Kortemme T 2024 Cell 187 526
 Google Scholar
Google Scholar
[102] Notin P, Rollins N, Gal Y, Sander C, Marks D 2024 Nat. Biotechnol. 42 216
 Google Scholar
Google Scholar
[103] Listov D, Goverde C A, Correia B E, Fleishman S J 2024 Nat. Rev. Mol. Cell Biol. 25 639
 Google Scholar
Google Scholar
[104] Ingraham J, Garg V K, Barzilay R, Jaakkola T 2019 Proceedings of the 33rd International Conference on Neural Information Processing Systems Vancouver, BC, Canada, December 8–14, 2019 p15820
[105] Dauparas J, Anishchenko I, Bennett N, Bai H, Ragotte R J, Milles L F, Wicky B I M, Courbet A, de Haas R J, Bethel N, Leung P J Y, Huddy T F, Pellock S, Tischer D, Chan F, Koepnick B, Nguyen H, Kang A, Sankaran B, Bera A K, King N P, Baker D 2022 Science 378 49
 Google Scholar
Google Scholar
[106] Hsu C, Verkuil R, Liu J, Lin Z, Hie B, Sercu T, Lerer A, Rives A 2022 bioRxiv: 2022.04.10.487779[Systems Biology]
[107] Sohl-Dickstein J, Weiss E A, Maheswaranathan N, Ganguli S 2015 arXiv: 1503.03585[cs.LG]
[108] Ho J, Jain A, Abbeel P 2020 Advances in Neural Information Processing Systems 33 6840
 Google Scholar
Google Scholar
[109] Watson J L, Juergens D, Bennett N R, et al 2023 Nature 620 1089
 Google Scholar
Google Scholar
[110] Song Y, Sohl-Dickstein J, Kingma D P, Kumar A, Ermon S, Poole B 2020 arXiv: 2011.13456[cs.LG]
[111] Lee J S, Kim J, Kim P M 2023 Nature Computational Science 3 382
 Google Scholar
Google Scholar
[112] Liu Y, Chen L, Liu H 2023 bioRxiv: 2023.11.18.567666 [Bioinformatics]
[113] Zheng Z, Deng Y, Xue D, Zhou Y, YE F, Gu Q 2023 arXiv: 2302.01649[cs.LG]
[114] Yang K K, Zanichelli N, Yeh H 2023 Protein Eng. Des. Sel. 36 gzad015
 Google Scholar
Google Scholar
[115] Kaplan J, McCandlish S, Henighan T, Brown T B, Chess B, Child R, Gray S, Radford A, Wu J, Amodei D 2020 arXiv: 2001.08361[cs.LG]
[116] He K, Chen X, Xie S, Li Y, Dollár P, Girshick R 2021 arXiv: 2111.06377[cs.CV]
[117] Chen T, Kornblith S, Norouzi M, Hinton G 2020 arXiv: 2002.05709[cs.LG]
[118] Wang Z, Wang Z, Srinivasan B, Ioannidis V N, Rangwala H, Anubhai R 2023 arXiv: 2310.03320[cs.LG]
[119] Von Rueden L, Mayer S, Beckh K, Georgiev B, Giesselbach S, Heese R, Kirsch B, Walczak M, Pfrommer J, Pick A, Ramamurthy R, Garcke J, Bauckhage C, Schuecker J 2021 IEEE Trans. Knowl. Data Eng. 35 614
[120] Bao L, Zhang X, Jin L, Tan Z J 2015 Chin. Phys. B 25 018703
 Google Scholar
Google Scholar
[121] Qiang X W, Zhang C, Dong H L, Tian F J, Fu H, Yang Y J, Dai L, Zhang X H, Tan Z J 2022 Phys. Rev. Lett. 128 108103
 Google Scholar
Google Scholar
[122] Dong H L, Zhang C, Dai L, Zhang Y, Zhang X H, Tan Z J 2024 Nucleic Acids Res. 52 2519
 Google Scholar
Google Scholar
Catalog
Metrics
- Abstract views: 7016
- PDF Downloads: 127
- Cited By: 0













 DownLoad:
DownLoad:
